Radiologia Brasileira - Publicação Científica Oficial do Colégio Brasileiro de Radiologia
AMB - Associação Médica Brasileira CNA - Comissão Nacional de Acreditação
 Vol. 54 nº 3 - Maio / Jun. of 2021
Vol. 54 nº 3 - Maio / Jun. of 2021
|
ARTIGOS ORIGINAIS
|
|
|
|
|
Autho(rs): Fernando Carrasco Ferreira Dionisio1,a; Larissa Santos Oliveira1,b; Mateus de Andrade Hernandes1,c; Edgard Eduard Engel1,d; Paulo Mazzoncini de Azevedo-Marques2,e; Marcello Henrique Nogueira-Barbosa2,f |
|
|
Descritores: Processamento de imagem assistida por computador; Sarcoma/diagnóstico por imagem; Neoplasias de tecidos moles/diagnóstico por imagem; Ressonância magnética; Reprodutibilidade dos testes. |
|
|
Resumo: INTRODUÇÃO
Os sarcomas de tecidos moles constituem um grupo heterogêneo de tumores malignos com ampla variedade histológica e prognóstica(1). Embora acometam os tecidos conjuntivos do corpo todo, a maioria deles se origina nas extremidades (59%), no tronco (19%), no retroperitônio (15%) ou na região da cabeça e pescoço (9%)(2,3). Os sarcomas de tecidos moles são responsáveis por aproximadamente 7% a 15% dos tumores malignos pediátricos e 1% dos tumores malignos nos adultos(2). A ressonância magnética (RM) é o método recomendado para avaliação dos tumores de tecidos moles pela excelente resolução de contraste, por não utilizar radiação ionizante e pela capacidade de aquisição de imagens em múltiplos planos(4). A radiômica é uma proposta de extrair grande quantidade de informações quantitativas dos exames de imagem disponíveis na rotina clínica e integrar modelos preditivos de diagnóstico, prognóstico e resposta terapêutica(5). As imagens médicas contêm informações mensuráveis que podem refletir a fisiopatologia da doença e que podem ser reveladas via análise quantitativa(6). Para aplicar a radiômica, o processo de segmentação é essencial, consistindo na marcação da região de interesse de onde as informações quantitativas serão extraídas para análise. Existem três tipos de segmentação: manual, semiautomática e automática. A segmentação manual consiste na segmentação da lesão ou da região anatômica de interesse utilizando interface gráfica, como um mouse ou uma caneta digital, por exemplo, com a qual o usuário marca os limites da lesão ou do órgão segmentado, processo que consome tempo e exige conhecimento da anatomia seccional nas imagens. A segmentação semiautomática consiste na utilização de softwares com algoritmos de segmentação que, a partir de informações fornecidas inicialmente pelo usuário de forma manual, delimitam a lesão. De forma geral, pode ser necessário fazer ajustes manuais após a obtenção desta seleção. A segmentação automática consiste na identificação completamente automatizada dos contornos da lesão pelo software, sem utilização de dados pré-fornecidos manualmente. Embora a segmentação automática aparente ser mais eficaz, principalmente considerando seu potencial para economia de tempo, este tipo de segmentação demanda recursos computacionais complexos e, em geral, não é simples obter resultados satisfatórios(7). Encontramos diversos estudos na literatura que utilizaram segmentação manual(8–15), semiautomática(16,17) e automática(18) de sarcomas de tecidos moles em RM. Em apenas um desses estudos foi realizada análise da similaridade interobservador(13), mas não encontramos estudos sobre a similaridade intraobservador. Mais importante, não encontramos, na literatura científica, estudos sobre a similaridade das segmentações obtidas pelos métodos de segmentação manual e semiautomático em sarcomas de partes moles, lacuna na literatura disponível que nos motivou a realizar o presente estudo. A avaliação dos métodos de segmentação manual e semiautomático e o potencial de poupar tempo de segmentação também justificaram nosso interesse pelo desenvolvimento deste estudo. Os objetivos deste estudo foram avaliar a similaridade entre as segmentações manual e semiautomática de sarcomas de tecidos moles nas imagens de RM e a similaridade intraobservador e interobservador das segmentações manuais. MATERIAIS E MÉTODOS O estudo foi aprovado pelo comitê de ética em pesquisa do hospital universitário, tendo sido dispensado o termo de consentimento livre e esclarecido em razão de o estudo ter utilizado imagens de forma retrospectiva. Todas as informações dos pacientes contidas nos arquivos DICOM das RMs foram anonimizadas utilizando o software KPACS (IMAGE Information Systems, Rostock, Alemanha). Para garantir a privacidade de cada paciente, os arquivos foram identificados por meio de números (por exemplo, paciente nº 1, paciente nº 2) e não pelo nome ou pelas iniciais do nome. Seleção dos casos e das imagens Todos os exames do presente estudo foram adquiridos em equipamento Philips Achieva 1.5 tesla (Philips Medical Systems, Eindhoven, Holanda). Foram selecionados casos desde janeiro de 2006 a janeiro de 2016 que estavam no sistema de informação radiológica de nossa instituição, pesquisando a palavra-chave “sarcoma” na conclusão dos relatórios de RM. Para a segmentação, foram selecionadas imagens no plano axial, ponderadas na sequência T2 com supressão de gordura. Foram utilizados tempo de repetição (TR) médio de 4316 ms (variando de 3489 a 4733 ms), tempo de eco (TE) médio de 55 ms (variando de 50 a 60 ms), matriz de 563 × 563 pixels (variando de 320 × 320 a 864 × 864 pixels), espessura do corte médio de 5,2 mm (variando de 3 a 7 mm) e resolução espacial média de 0,571 mm/pixel (variando de 0,234 a 0,742 mm/pixel). Em um dos casos (um lipossarcoma bem diferenciado) foi usada a sequência T1 sem supressão de gordura, com TR de 352 ms, TE de 10 ms, matriz de 480 × 480 pixels, espessura do corte de 6,0 mm e resolução espacial de 0,686 mm/pixel. Os critérios para a seleção dos casos incluídos no estudo foram: confirmação diagnóstica de sarcoma de tecido moles por estudo histopatológico no sistema de informações hospitalares da instituição; localização do tumor no esqueleto apendicular (membros superiores ou inferiores); disponibilidade de imagens de exame de RM no formato DICOM, realizado em nossa instituição, previamente a tratamento ou intervenção diagnóstico-terapêutica (biópsia, cirurgia, quimioterapia ou radioterapia). Dessa seleção, foram excluídos os casos em que artefatos de movimento prejudicaram a análise das imagens. A Figura 1 resume o fluxo de etapas de formação da casuística. 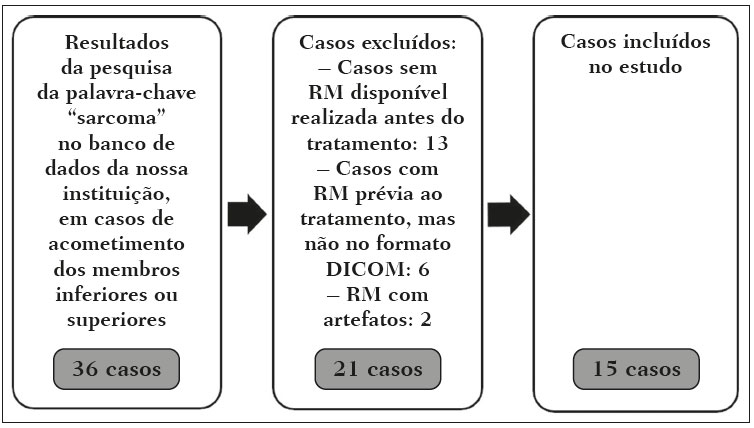 Figura 1. Fluxograma da seleção dos casos incluídos no estudo. Análise e segmentação da RM As segmentações manual e semiautomática foram realizadas no software 3D Slicer (versão 4.6.2), de acesso livre para processamento de imagens médicas, disponível em https://www.slicer.org. A segmentação semiautomática foi realizada com a ferramenta GrowCut disponível no software 3D Slicer. As segmentações foram realizadas em imagens de RM adquiridas no plano axial ponderadas em T2 (T2WI) com supressão de gordura. Em apenas um caso (lipossarcoma), imagens ponderadas em T1 (T1WI) sem supressão de gordura no plano axial foram utilizadas para auxiliar no processo de segmentação. Três radiologistas realizaram a segmentação manual dos sarcomas de forma independente e cegos em relação às demais segmentações e resultados histopatológicos. Dois radiologistas (radiologista 1 e radiologista 2) realizaram as segmentações durante o fellow de especialização em radiologia musculoesquelética, enquanto o radiologista 3 tinha cinco anos de experiência em radiologia musculoesquelética. O radiologista 1 segmentou manualmente todos os casos uma segunda vez após um mês de intervalo, para avaliação da similaridade intraobservador. A avaliação da similaridade interobservador foi realizada comparando as segmentações dos três radiologistas. Outra análise foi realizada para comparar a segmentação manual e a segmentação semiautomática realizada pelo radiologista 1. O radiologista 1 realizou segmentação semiautomática dois meses após a segunda segmentação manual, para comparar ambas. A segmentação manual foi realizada pela demarcação manual dos limites do tumor em cada corte seccional, em todos os cortes seccionais onde havia presença de tecido tumoral (Figura 2), de acordo com procedimentos em estudos prévios(13,19–23). 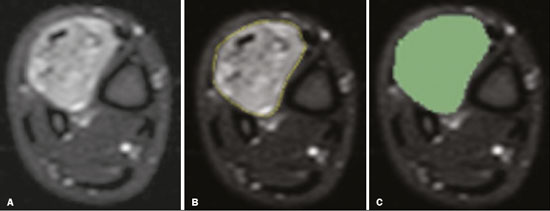 Figura 2. Etapas da segmentação manual. A: Identificação do tumor. B: Delimitação dos limites do tumor (linha amarela). C: Criando a área de interesse segmentada em um corte do tumor (área em verde). No processo de segmentação semiautomática (Figura 3) com a ferramenta GrowCut, o usuário identifica grosseiramente regiões no interior do tecido tumoral e dos tecidos adjacentes (não tumorais). Após este procedimento, o software atua demarcando um volume de interesse segmentado. É possível efetuar correções manuais nos contornos obtidos pelo software. Existem diversos algoritmos para segmentação semiautomática, e nosso método condiz com o proposto por Egger et al.(19,20) utilizando a ferramenta GrowCut disponível no software 3D Slicer. 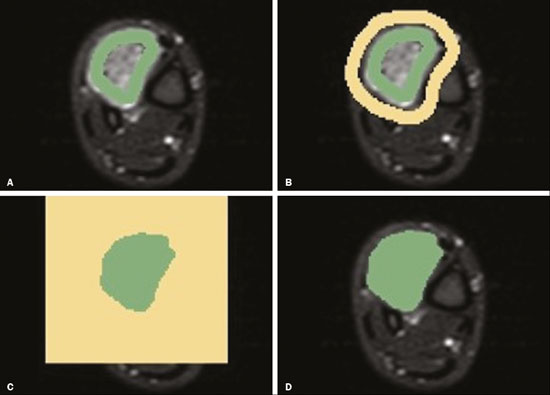 Figura 3. Etapas da segmentação semiautomática. A: Marcação de parte da região interna do tumor (em verde). B: Marcação de parte da região externa adjacente ao tumor (em amarelo). C: Segmentação fornecida pela ferramenta GrowCut da área interna (em verde) e externa (em amarelo) do tumor. D: Somente a segmentação a área interna do tumor foi selecionada para criar a área de interesse. No presente estudo, para ambos os métodos de segmentação (manual e semiautomático), o volume de interesse segmentado representou a extensão total do tumor. O volume não incluiu a área de edema peritumoral na segmentação. Outro critério na segmentação foi incluir estruturas neurovasculares ou tendíneas na área segmentada quando essas estruturas anatômicas estavam envolvidas circunferencialmente (360 graus) pela neoplasia no plano axial. Neste estudo, escolhemos a supressão de gordura no plano axial T2WI como a sequência padrão para realizar segmentações, porque esta sequência estava disponível em todos os casos. Em casos em que a sequência axial T2WI com supressão apresentava baixo contraste tecidual entre o tumor e os tecidos circundantes, a sequência T1WI estava disponível e foi utilizada para auxiliar no processo de segmentação. O tempo necessário para as segmentações manual e semiautomática pelo radiologista 1 foi registrado para posterior comparação, utilizando um cronômetro digital controlado por outro pesquisador que não estava realizando a segmentação. Na contagem de tempo da segmentação manual, as imagens foram abertas no software 3D Slicer e uma rápida visualização geral do tumor era realizada. A aferição do tempo era iniciada imediatamente antes de iniciar o processo de demarcação dos contornos tumorais, sendo encerrada após a demarcação dos contornos tumorais no último corte seccional com presença de tecido tumoral. Na segmentação semiautomática, as imagens foram abertas no software 3D Slicer e uma rápida visualização geral do tumor era realizada, sendo identificado o corte seccional central do tumor após contagem do número de cortes totais em que era observada a presença de tecido tumoral. A aferição do tempo era iniciada imediatamente antes do início da demarcação manual do tumor e do tecido externo ao tumor neste corte e era encerrada após serem realizadas as correções manuais sobre o mapa de segmentação gerado pelo algoritmo GrowCut. Tal padronização seguiu estudos de segmentação prévios com o software 3D Slicer e a ferramenta GrowCut em outros tipos histológicos de tumores(19,20). Análise estatística O coeficiente de similaridade Dice (CSD) e a distância de Hausdorff (DH) foram utilizados para avaliar a similaridade entre as segmentações manual e semiautomática e a similaridade intraobservador e interobservador das segmentações manuais. Tanto o CSD quanto a DH são fornecidos pelo software 3D Slicer a partir da comparação das segmentações realizadas. O CSD mede a sobreposição espacial e pode servir como uma métrica para validação da concordância entre duas segmentações, variando de 0 a 1. O valor “0” significa que não há sobreposição espacial e o valor “1” indica que há sobreposição total das segmentações. Neste estudo, adotamos os seguintes critérios para interpretação do CSD: baixa similaridade, CSD < 0,7; boa similaridade, 0,7 ≤ CSD < 0,8(24–27); alta similaridade, CSD ≥ 0,8(28). A DH entre dois volumes segmentados é definida em termos da distância euclidiana entre seus voxels de fronteira. Por exemplo, considerando os voxels de limite de segmentação semiautomática e os voxels de limite da segmentação manual, a DH máxima é definida como a distância euclidiana máxima entre qualquer um dos pontos no primeiro grupo e os pontos do segundo grupo(20). Também foram feitas comparações dos volumes obtidos nas segmentações. Médias, desvios-padrão (DPs) e coeficientes de correlação intraclasse (CCIs) foram obtidos a partir dos volumes segmentados. Comparações estatísticas de volumes foram realizadas em diferentes situações para verificar se havia diferença estatisticamente significante entre eles. Os tempos para as segmentações manual e semiautomática realizadas pelo radiologista 1 foram comparados e as médias e DPs foram obtidos, tendo-se aplicado o teste de Wilcoxon para verificar diferenças estatísticas no tempo de execução da segmentação entre esses métodos. Adicionalmente, verificamos a correlação entre o tempo de segmentação e o volume tumoral a ser segmentado, utilizando o coeficiente de correlação de Spearman. RESULTADOS Após a pesquisa em nosso sistema de informação radiológica pelo termo “sarcoma”, obtivemos 36 exames nos quais esses tumores estavam localizados nas extremidades. Após análise dos exames e pesquisa de informações clínicas no sistema de informações hospitalares, constatamos que 13 exames não dispunham de RM realizada previamente a qualquer intervenção terapêutica ou diagnóstica invasiva. Dos 23 exames restantes, 6 não estavam disponíveis no formato DICOM e 2 exames apresentaram artefatos, sendo excluídos de nossa casuística. Ao final da seleção dos casos em nosso estudo, obtivemos exames de 15 pacientes, sendo 8 do sexo feminino e 7 do sexo masculino, com idades entre 6 e 91 anos (idade média de 50,8 anos). Todos os pacientes tiveram diagnóstico confirmado de sarcoma de tecidos moles com sete subtipos histológicos diferentes, sendo o mais frequente o lipossarcoma, presente em cinco dos pacientes. A localização dos tumores nos membros foi variável, sendo a coxa o local mais acometido (cinco casos). A Tabela 1 expõe os dados epidemiológicos dos casos deste estudo, incluindo os subtipos histopatológicos dos tumores.  Segmentação manual – similaridade intraobservador As duas segmentações realizadas pelo radiologista 1 foram comparadas e o CSD variou de 0,849 a 0,979 e a DH variou de 3,53 mm a 20,96 mm. A comparação entre os volumes dessas duas segmentações manuais resultoub em CCI de 0,999. A Tabela 2 resume a análise de comparação intraobservador das segmentações. O tempo requerido para a segmentação manual dos casos variou de 2 a 32 minutos, sendo a média de 12,4 minutos, com DP de ± 7,73 minutos. 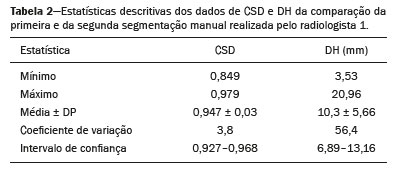 A correlação de Spearman obtida na comparação entre os volumes tumorais e o tempo de segmentação foi de 0,59, com valor de p = 0,020, indicando que a associação entre as duas variáveis poderia ser considerada estatisticamente significante. Segmentação manual – similaridade interobservador As segmentações manuais realizadas pelos três radiologistas foram comparadas entre si. O CSD obtido a partir da comparação entre as segmentações manuais realizadas pelos radiologistas 1 e 2 variou de 0,781 a 0,973 e a DH variou de 5,83 mm a 27,81 mm. A comparação das segmentações realizadas pelos radiologistas 1 e 3 resultou em CSD variando de 0,741 a 0,972 e DH variando de 8,08 mm a 61,84 mm. As Tabelas 3 e 4 resumem a análise da comparação interobservadores das segmentações. 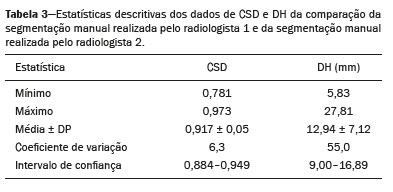 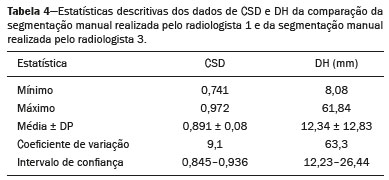 A comparação entre os volumes obtidos na segmentação manual realizada pelos radiologistas 1 e 2 resultou em CCI de 0,9976. Resultados semelhantes foram obtidos a partir da comparação entre os volumes obtidos nas segmentações manuais realizadas pelos radiologistas 1 e 3, com CCI de 0,9927. Segmentação semiautomática A comparação entre segmentação manual e segmentação semiautomática realizada pelo radiologista 1 resultou em CSD variando de 0,871 a 0,973 e DH variando de 5,43 mm a 31,75 mm. A comparação entre os volumes obtidos nas segmentações manual e semiautomática resultou em CCI de 0,9990. A Tabela 5 resume a análise entre as segmentações manual e semiautomática. O tempo requerido para a segmentação semiautomática dos casos variou de 7 a 34 minutos, sendo a média de 13,8 minutos, com DP de ± 7,23 minutos. 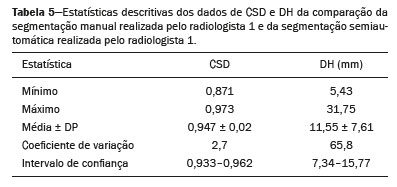 A correlação de Spearman obtida na comparação entre os volumes tumorais e o tempo de segmentação foi de 0,32, com valor de p = 0,25, indicando que a associação entre as duas variáveis não pode ser considerada estatisticamente significante. Comparação do tempo de segmentação entre os métodos manual e semiautomático Um tempo menor na segmentação manual foi obtido em 9 dos 15 casos segmentados, comparando o tempo de segmentação por ambos os métodos, mas a diferença não foi estatisticamente significante ao ser avaliada pelo teste de Wilcoxon (p > 0,05). Na Tabela 6 detalhamos os volumes tumorais segmentados e os tempos de segmentação para as segmentações manual e semiautomática realizadas pelo radiologista 1. DISCUSSÃO Nossos resultados mostram alta similaridade na comparação entre a segmentação semiautomática e a segmentação manual de sarcomas de tecidos moles em imagens de RM, no entanto, não se observou redução significativa de tempo esperada para o método semiautomático. Houve alta similaridade intraobservador (CSD ≥ 0,8) e boa similaridade interobservador das segmentações manuais (0,7 ≤ CSD < 0,8). A comparação entre os volumes segmentados mostrou grande concordância, reforçando a boa reprodutibilidade entre as segmentações pelos dois métodos. Nossos resultados são consistentes com a literatura, que indica algo grau de similaridade entre as segmentações manual e semiautomática de outras neoplasias em imagens de RM, como, por exemplo, no caso de glioblastomas multiformes(19), adenomas hipofisários(20) e carcinomas hepatocelulares(27). Há convergência dos nossos resultados com resultados anteriores da literatura, indicando alta similaridade interobservador na segmentação manual de sarcomas de partes moles em imagens de RM, como demonstrado no estudo de Peeken et al.(13), em que foi obtido um coeficiente de similaridade ainda maior, com CSD de 0,91 (DP: ± 0.069). Porém, nesse estudo(13), os autores não realizaram comparação da segmentação manual com a semiautomática, não avaliaram a reprodutibilidade dos volumes das segmentações, e incluíram avaliação de sarcomas abdominais ou retroperitoniais. A diferença de contraste tecidual de diversas sequências de RM pode impactar na segmentação do tumor, com potencial para influenciar na reprodutibilidade do método. No caso de sarcomas de tecidos moles, essa escolha não é uma tarefa fácil, por causa da variedade de subtipos histológicos e suas características de imagem potencialmente heterogêneas. A literatura aponta uma variabilidade das sequências escolhidas para segmentação e análise radiômica desses tumores(8–16). Vários estudos recentes que aplicaram radiômica em sarcomas de tecidos moles(8,12–15) utilizaram a segmentação em imagens ponderadas na sequência T2WI de forma semelhante ao nosso estudo, al-guns deles também efetuando segmentações em outras sequências para análises específicas. Em alguns estudos de análise radiômica de sarcomas de tecidos moles em RM, apesar da segmentação ser obtida em uma sequência específica, outras sequências estavam disponíveis para an´liseanálise(9,11,12). Escolhemos a sequência T2WI no plano axial porque era a única que estava disponível em todos os casos e também por possibilitar alto contraste tecidual entre a neoplasia e os tecidos moles adjacentes. Em apenas um caso de lipossarcoma bem diferenciado utilizamos também imagens T1WI sem supressão de gordura durante a segmentação, para ajudar no delineamento do tumor, por se apresentar mais conspícuo na sequência T1WI sem supressão de gordura. Apesar de cinco casos de nosso estudo representarem lipossarcomas, apenas em um único caso o lipossarcoma bem diferenciado não apresentava limites suficientemente distintos para a delimitação nas sequências ponderadas em T2WI com supressão de gordura. Os demais apresentavam predomínio de tecido mixoide ou eram de alto grau e tinham seus contornos suficientemente delimitados para que fosse realizada a segmentação na sequência T2WI. Hipoteticamente, a sequência T1WI sem supressão de gordura pode trazer mais benefício na segmentação de lipossarcomas, especialmente os mais bem diferenciados histologicamente, mas nosso estudo não possibilita comparar especificamente a influência da utilização de diferentes sequências nos resultados de similaridade das segmentações. Um estudo que analisou especi ficamente a diferenciação entre lipomas e lipossarcomas realizou a segmentação em imagens T1WI, ressaltando a importância desta sequência na avaliação de tumores de linhagem lipomatosa, porém, não foi avaliada a reprodutibilidade da segmentação em si(16). Vallières et al.(8) realizaram segmentação manual delimitando os contornos de sarcomas de tecidos moles, e nos casos em que o edema perilesional era visível realizaram segmentação adicional incorporando a área do edema, não sendo avaliada, entretanto, a reprodutibilidade dessa segmentação no estudo. Como mencionado em nosso método, excluímos o edema peritumoral da segmentação dos nossos casos. A importância potencial da área de edema se relaciona a possível presença de células tumorais nessa região(29). Na prática clínica, no entanto, o edema perilesional nem sempre é ressecado cirurgicamente(29). Múltiplos estudos que realizaram análise radiômica em casos de sarcomas de tecidos moles também não incluíram o edema peritumoral(9–13,15), o que reforça a potencial aplicação de nossos resultados. Ainda não há estudos específicos que avaliem a importância do edema peritumoral em casos de sarcomas ósseos na análise radiômica, tampouco estudos que tenham avaliado a reprodutibilidade da segmentação desse edema. Como o edema costuma apresentar aspecto mal delimitado, presumivelmente pode representar fator desafiador para a obtenção de alto grau de similaridade na segmentação. Em casos de tumores cerebrais(30) e meningiomas cranianos(31), foi demonstrado alto grau de similaridade da segmentação do edema tumoral utilizando métodos semiautomáticos, entretanto, não encontramos estudos na literatura científica que tenham efetuado especificamente a avaliação da similaridade da segmentação do edema peritumoral em casos de sarcomas de partes moles. Sugerimos estudos futuros para preencher esta lacuna da literatura em relação à segmentação do edema peritumoral nos sarcomas de tecidos moles. O tempo para segmentação manual pode ser influenciado por vários fatores, sendo os principais o volume e a delimitação das margens do tumor(32,33). Quanto maior o tumor, maior o número de cortes seccionais serão analisados e segmentados, com tendência a aumentar o tempo necessário para segmentação. Nossos resultados mostraram que houve correlação estatística entre o volume do tumor e o tempo de segmentação manual, tendo o tempo de segmentação manual sido maior para tumores com maior volume. A dificuldade em delimitar o tumor é maior se suas margens forem mal delimitadas e com pouco contraste tecidual entre o tumor e os tecidos adjacentes, constituindo um desafio no caso da segmentação de neoplasias, especialmente as que apresentam com aspecto heterogêneo nos exames de imagem(6,7). Em nossa casuística tivemos casos em que as margens do sarcoma eram mal delimitadas e, por este motivo, a segmentação semiautomática extrapolou os limites da segmentação incluindo tecidos que não pertenciam ao tumor. Estes casos aumentaram o tempo de segmentação por demandar correção dos contornos do tumor e o tempo gasto para correção anulou o potencial ganho de tempo que geralmente é obtido com o uso da segmentação semiautomática. Apesar da potencial variabilidade entre segmentações manuais realizadas por especialistas, estas são consideradas o padrão ouro disponível para a comparação entre métodos automático e semiautomático(34). Nos casos em que há pouco contraste entre a neoplasia e os tecidos vizinhos, a segmentação semiautomática também tem maior probabilidade de obter resultados que não representam a delimitação correta do sarcoma, o que requer muitos ajustes manuais do usuário, aumentando o tempo total de segmentação. Esta situação ocorreu, por exemplo, em um caso do nosso estudo, com diagnóstico de fibrossarcoma pouco diferenciado envolvendo a região da mão, com lesão infiltrativa e de margens pouco delimitadas. A correlação obtida entre o volume tumoral e o tempo da segmentação manual dos tumores confirma que o volume tumoral é um fator que influencia neste método de segmentação, sendo necessário mais tempo para segmentar tumores maiores. No caso da segmentação semiautomática, o tempo de segmentação não apresentou correlação estatística com o volume do tumor e, portanto, outros fatores devem ter importância significativa ao influenciar no tempo de segmentação quando o modelo semiautomático é adotado. Conforme já mencionado, acreditamos que no caso da segmentação semiautomática, os tumores de contornos mal delimitados demandam tempo maior de correção pelo radiologista. É reconhecido na literatura científica que algoritmos baseados em crescimento contínuo de regiões marcadas manualmente tendem a gerar hipersegmentação, ou seja, segmentações que incluem zonas além dos limites reais da estrutura a ser segmentada, especialmente para lesões ou estruturas de limites mal definidos(7). Não encontramos estudos que fizeram a comparação do tempo de segmentação de tumores de contornos mal definidos e tumores bem delimitados. Sugerimos que esta seja uma possibilidade interessante para pesquisas futuras. Não identificamos estudos na literatura padronizando a mensuração de tempos de segmentação em casos de sarcomas de tecido moles. A ausência de diferença estatística significativa para o tempo médio de segmentação comparando os métodos semiautomático e manual em nosso estudo difere dos resultados de estudos encontrados para outros tipos de neoplasias. Egger et al.(19,20) demonstraram que a segmentação semiautomática seria um método mais rápido que a segmentação manual em glioblastomas e adenomas da hipófise, que são tumores do sistema nervoso central. Dionisio et al.(23) descrevem vantagem com economia de tempo na segmentação semiautomática de sarcomas ósseos em imagens de RM em relação à segmentação manual. Presumimos que nossos resultados não reproduziram os resultados de estudos com tumores cerebrais e sarcomas ósseos, em razão da grande heterogeneidade tecidual dos sarcomas de tecidos moles incluídos em nosso estudo, quando comparados a outros tipos de tumores; no nosso estudo, os métodos de segmentação semiautomática apresentaram a tendência de demandar maior correção manual no resultado da segmentação semiautomática obtida pelo software, conforme discussão da literatura prévia(32). O algoritmo de segmentação utilizado pela ferramenta GrowCut se baseia em buscar semelhanças nos tons de cinza entre pixels na borda da lesão e pixels externos, mas adjacentes à borda. Quando o tumor é heterogêneo, há maior probabilidade de que a periferia da neoplasia apresente pixels com intensidade de sinal semelhantes aos tecidos não pertencentes ao tumor, estendendo a segmentação semiautomática para regiões externas que não pertencem à lesão neoplásica. Assim, para alguns tumores de tecidos moles pode ser preciso um tempo maior, pela necessidade de correção manual para se chegar à segmentação final correta do tumor. Não existe algoritmo de segmentação universalmente aceito e satisfatório para todos os tipos de imagens médicas(7). Em nossa amostragem, obtivemos tempos de segmentação manual inferiores ao tempo de segmentação semiautomática em 9 dos 15 casos. Eventualmente, apesar de nossos resultados não terem demonstrado diferença estatística significativa do tempo despendido entre as segmentações manual e semiautomática, presumivelmente poderia haver o benefício da redução do tempo com a segmentação semiautomática em casos de sarcomas de maiores dimensões, mas com contornos bem delimitados. Nossa casuística não permitiu avaliação estatística robusta para confirmar esta hipótese. Sendo assim, pesquisas futuras precisam averiguar esta hipótese. Nosso estudo possui limitações que merecem ser mencionadas. Em primeiro lugar, é retrospectivo e inclui um número relativamente pequeno de casos. Nossa instituição é um centro de referência para o tratamento de sarcomas e de outros tumores de tecidos moles; no entanto, vários pacientes que são encaminhados de outros serviços para tratamento já realizaram estudos de RM previamente em outras instituições, e esses casos não foram incluídos em nossa casuística. Outra limitação é o fato de um único radiologista ter realizado a segmentação semiautomática e somente este radiologista ter tido o tempo de segmentação mensurado para comparação das diferentes técnicas de segmentação. O CSD é uma ferramenta estatística utilizada para avaliar o grau de semelhança entre duas amostras ou dois conjuntos de dados, possibilitando identificar a sobreposição entre as duas segmentações e sendo atualmente a forma mais utilizada para mensurar a similaridade entre diferentes segmentações realizadas em imagens médicas. O coeficiente pode, portanto, ser utilizado para inferir a similaridade entre as segmentações entre diferentes examinadores ou para calcular a similaridade entre a segmentação realizada com uma técnica considerada padrão ouro e outra técnica de segmentação. Não existe consenso na literatura sobre padronização específica para interpretação dos resultados de similaridade pelo CSD em estudos de segmentação(35). Alguns autores utilizaram, em seus estudos, o valor do CSD de 0,70 como limite inferior para considerar uma boa similaridade entre as segmentações(24–27). Zijdenbos et al.(26) e Fleiss(36) consideraram o CSD como um tipo especial de estatística kappa e definiram que valores do CSD superiores a 0,7 seriam equivalentes a kappa acima de 0,75 e que podem ser considerados como similaridade excelente(37). Por outro lado, Fontina et al.(35) advertem que os valores limites definidos pela estatística kappa por Landis et al.(38) foram originalmente introduzidos para dados categóricos e que a utilização dos mesmos parâmetros de interpretação da estatística kappa para a análise do CSD entre segmentações poderia superestimar a similaridade. Mattiucci et al.(28) sugerem um valor de CSD de 0,8 como limite inferior para considerar que foi obtida alta similaridade entre segmentações de diferentes médicos especialistas. Nossos resultados na avaliação da similaridade na segmentação manual e semiautomática de sarcomas de tecidos moles podem ser importantes para o contexto da radiômica com base em imagens de RM. Obtivemos alta similaridade intraobservador (CSD ≥ 0,8) e boa similaridade interobservador das segmentações manuais (0,7 ≤ CSD < 0,8) desses tumores. Sugerimos estudos futuros para avaliar ferramentas de segmentação semiautomáticas úteis para reduzir tempo e esforço no processo de segmentação, uma vez que com a ferramenta utilizada não conseguimos obter redução do tempo de segmentação no método semiautomático. CONCLUSÃO Os resultados obtidos mostraram alta similaridade entre as segmentações realizadas pelo método semiautomático quando comparadas às segmentações realizadas pelo método manual, apesar de o método semiautomático não ter uma diferença estatística significativa no tempo de segmentação em relação ao método manual neste estudo. Também obtivemos alta similaridade intraobservador e boa similaridade interobservadores na segmentação manual utilizando o software 3D Slicer. REFERÊNCIAS 1. Coindre JM. Grading of soft tissue sarcomas: review and update. Arch Pathol Lab Med. 2006;130:1448–53. 2. Morrison BA. Soft tissue sarcomas of the extremities. Proc (Bayl Univ Med Cent). 2003;16:285–90. 3. Cormier JN, Pollock RE. Soft tissue sarcomas. CA Cancer J Clin. 2004;54:94–109. 4. Nystrom LM, Reimer NB, Reith JD, et al. Multidisciplinary management of soft tissue sarcoma. ScientificWorldJournal. 2013;2013: 852462. 5. Koenigkam-Santos M, Ferreira Júnior JR, Wada DT, et al. Artificial intelligence, machine learning, computer-aided diagnosis, and radiomics: advances in imaging towards to precision medicine. Radiol Bras. 2019;52:387–96. 6. Gillies RJ, Kinahan PE, Hricak H. Radiomics: images are more than pictures, they are data. Radiology. 2016;278:563–77. 7. Kumar V, Gu Y, Basu S, et al. QIN Radiomics: the process and the challenges. Magn Reson Imaging. 2012;30:1234–48. 8. Vallières M, Freeman CR, Skamene SR, et al. A radiomics model from joint FDG-PET and MRI texture features for the prediction of lung metastases in soft-tissue sarcomas of the extremities. Phys Med Biol. 2015;60:5471–96. 9. Xie H, Hu J, Zhang X, et al. Preliminary utilization of radiomics in differentiating uterine sarcoma from atypical leiomyoma: comparison on diagnostic efficacy of MRI features and radiomic features. Eur J Radiol. 2019;115:39–45. 10. Corino VDA, Montin E, Messina A, et al. Radiomic analysis of soft tissues sarcomas can distinguish intermediate from high-grade lesions. J Magn Reson Imaging. 2018;47:829–40. 11. Crombé A, Le Loarer F, Sitbon M, et al. Can radiomics improve the prediction of metastatic relapse of myxoid/round cell liposarcomas? Eur Radiol. 2020;30:2413–24. 12. Crombé A, Fadli D, Buy X, et al. High-grade soft-tissue sarcomas: can optimizing dynamic contrast-enhanced MRI postprocessing improve prognostic radiomics models? J Magn Reson Imaging. 2020; 52:282–97. 13. Peeken JC, Spraker MB, Knebel C, et al. Tumor grading of soft tissue sarcomas using MRI-based radiomics. EBioMedicine. 2019; 48:332–40. 14. Tagliafico AS, Bignotti B, Rossi F, et al. Local recurrence of soft tissue sarcoma: a radiomic analysis. Radiol Oncol. 2019;53:300–6. 15. Wang H, Chen H, Duan S, et al. Radiomics and machine learning with multiparametric preoperative MRI may accurately predict the histopathological grades of soft tissue sarcomas. J Magn Reson Imaging. 2020;51:791–7. 16. Malinauskaite I, Hofmeister J, Burgermeister S, et al. Radiomics and machine learning differentiate soft-tissue lipoma and liposarcoma better than musculoskeletal radiologists. Sarcoma. 2020; 2020:7163453. 17. Monsky WL, Jin B, Molloy C, et al. Semi-automated volumetric quantification of tumor necrosis in soft tissue sarcoma using contrast- enhanced MRI. Anticancer Res. 2012;32:4951–61. 18. Farhidzadeh H, Chaudhury B, Zhou M, et al. Prediction of treatment outcome in soft tissue sarcoma based on radiologically defined habitats. Proc SPIE Medical Imaging. 2015;9414. 19. Egger J, Kapur T, Fedorov A, et al. GBM volumetry using the 3D Slicer medical image computing platform. Sci Rep. 2013;3:1364. 20. Egger J, Kapur T, Nimsky C, et al. Pituitary adenoma volumetry with 3D Slicer. PLoS One. 2012;7:e51788. 21. Velazquez ER, Parmar C, Jermoumi M, et al. Volumetric CT-based segmentation of NSCLC using 3D-Slicer. Sci Rep. 2013;3:3529. 22. Zou KH, Warfield SK, Bharatha A, et al. Statistical validation of image segmentation quality based on a spatial overlap index. Acad Radiol. 2004;11:178–89. 23. Dionísio FCF, Oliveira LS, Hernandes MA, et al. Manual and semiautomatic segmentation of bone sarcomas on MRI have high similarity. Braz J Med Biol Res. 2020;53:e8962. 24. Gambacorta MA, Valentini C, Dinapoli N, et al. Clinical validation of atlas-based auto-segmentation of pelvic volumes and normal tissue in rectal tumors using auto-segmentation computed system. Acta Oncol. 2013;52:1676–81. 25. Anders LC, Stieler F, Siebenlist K, et al. Performance of an atlasbased autosegmentation software for delineation of target volumes for radiotherapy of breast and anorectal cancer. Radiother Oncol. 2012;102:68–73. 26. Zijdenbos AP, Dawant BM, Margolin RA, et al. Morphometric analysis of white matter lesions in MR images: method and validation. IEEE Trans Med Imaging. 1994;13:716–24. 27. Tacher V, Lin M, Chao M, et al. Semiautomatic volumetric tumor segmentation for hepatocellular carcinoma: comparison between C-arm cone beam computed tomography and MRI. Acad Radiol. 2013;20:446–52. 28. Mattiucci GC, Boldrini L, Chiloiro G, et al. Automatic delineation for replanning in nasopharynx radiotherapy: what is the agreement among experts to be considered as benchmark? Acta Oncol. 2013;52:1417–22. 29. White LM, Wunder JS, Bell RS, et al. Histologic assessment of peritumoral edema in soft tissue sarcoma. Int J Radiat Oncol Biol Phys. 2005;61:1439–45. 30. Xie K, Yang J, Zhang ZG, et al. Semi-automated brain tumor and edema segmentation using MRI. Eur J Radiol. 2005;56:12–9. 31. Latini F, Larsson EM, Ryttlefors M. Rapid and accurate MRI segmentation of peritumoral brain edema in meningiomas. Clin Neuroradiol. 2017;27:145–52. 32. Rizzo S, Botta F, Raimondi S, et al. Radiomics: the facts and the challenges of image analysis. Eur Radiol Exp. 2018;2:36. 33. Deniz CM, Xiang S, Hallyburton RS, et al. Segmentation of the proximal femur from MR images using deep convolutional neural networks. Sci Rep. 2018;8:16485. 34. Stroom J, Blaauwgeers H, van Baardwijk A, et al. Feasibility of pathology-correlated lung imaging for accurate target definition of lung tumors. Int J Radiat Oncol Biol Phys. 2007;69:267–75. 35. Fotina I, Lütgendorf-Caucig C, Stock M, et al. Critical discussion of evaluation parameters for inter-observer variability in target definition for radiation therapy. Strahlenther Onkol. 2012;188:160–7. 36. Fleiss JL. Statistical methods for rates and proportions. 2nd ed. New York, NY: John Wiley; 1981. 37. Langmack KA, Perry C, Sinstead C, et al. The utility of atlasassisted segmentation in the male pelvis is dependent on the interobserver agreement of the structures segmented. Br J Radiol. 2014;87:20140299. 38. Landis JR, Koch GG. The measurement of observer agreement for categorical data. Biometrics. 1977;33:159–74. 1. Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (HCFMRP-USP), Ribeirão Preto, SP, Brasil 2. Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP-USP), Ribeirão Preto, SP, Brasil a. https://orcid.org/0000-0002-3681-9961 b. https://orcid.org/0000-0003-4572-9447 c. https://orcid.org/0000-0002-2244-5532 d. https://orcid.org/0000-0002-4047-2796 e. https://orcid.org/0000-0002-7271-2774 f. https://orcid.org/0000-0002-7436-5315 Correspondência: Dr. Fernando Carrasco Ferreira Dionisio Hospital das Clínicas – FMRP-USP. Avenida Bandeirantes, 3900, Monte Alegre Ribeirão Preto, SP, Brasil, 14048-900 E-mail: fcf.dionisio@yahoo.com.br Recebido para publicação em 24/3/2020 Aceito, após revisão, em 24/7/2020 |
|
Av. Paulista, 37 - 7° andar - Conj. 71 - CEP 01311-902 - São Paulo - SP - Brazil - Phone: (11) 3372-4544 - Fax: (11) 3372-4554