Radiologia Brasileira - Publicação Científica Oficial do Colégio Brasileiro de Radiologia
AMB - Associação Médica Brasileira CNA - Comissão Nacional de Acreditação
 Vol. 52 nº 6 - Nov. / Dez. of 2019
Vol. 52 nº 6 - Nov. / Dez. of 2019
|
ARTIGO DE REVISÃO
|
|
|
|
|
Autho(rs): Marcel Koenigkam Santos1,a; José Raniery Ferreira Júnior2,3,b; Danilo Tadao Wada1,c; Ariane Priscilla Magalhães Tenório3,d; Marcello Henrique Nogueira Barbosa3,e; Paulo Mazzoncini de Azevedo Marques3,f |
|
|
Descritores: Inteligência artificial; Aprendizado de máquina; Diagnóstico auxiliado por computador; Radiômica. |
|
|
Resumo: INTRODUÇÃO
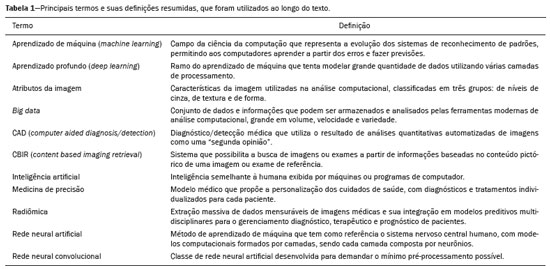
A disciplina de radiologia e diagnóstico por imagem evoluiu sobremaneira nos últimos anos. As imagens radiológicas podem ser extremamente complexas e a análise de exames com centenas de imagens, como a tomografia computadorizada (TC) e a ressonância magnética (RM), é um desafio reconhecido mesmo para especialistas experientes(1–3). As dificuldades vêm aumentado nos últimos anos, com o aumento exponencial do número de exames realizados, a subespecialização das disciplinas médicas e a maior acurácia dos métodos, tornando um desafio para o médico radiologista “saber tudo sobre todos exames e regiões”. Além disso, os exames de imagem deixaram de ser somente qualitativos e diagnósticos e passaram a fornecer informações quantitativas e de gravidade de doença, identificando biomarcadores prognósticos e de resposta ao tratamento(4–6). Essas mudanças têm se destacado principalmente na oncologia, mostrando que as informações presentes nos exames de imagem são capazes de ir muito além de dizer se uma lesão é benigna ou maligna, podendo inferir o tipo histológico do tumor, o estadiamento, a presença de mutações, a chance de resposta ao tratamento, de recorrência e a sobrevida do paciente(7–9). Sistemas computadorizados de auxílio diagnóstico vêm sendo desenvolvidos com o objetivo de melhorar a acurácia dos exames, a consistência na interpretação de imagens médicas, a avaliação prognóstica e o suporte à decisão terapêutica. Essas ferramentas possuem potencial enorme, porém, há ainda limitações para seu uso na rotina clínica. Com o advento da inteligência artificial e do big data, caminhamos para a redução dessas limitações, homogeneização e expansão do uso dessas ferramentas no dia-a-dia dos médicos, tornando cada paciente único, levando a radiologia ao encontro do conceito de abordagem multidisciplinar e medicina de precisão(10–14). Neste artigo serão abordados os principais aspectos das ferramentas computacionais atualmente disponíveis para análise das imagens médicas, discutindo também o impacto do desenvolvimento da inteligência artificial e o papel da imaginologia na medicina de precisão. A Tabela 1 apresenta os principais termos e suas definições resumidas, que serão utilizados ao longo deste texto. PRINCÍPIOS DA ANÁLISE COMPUTADORIZADA DE IMAGENS MÉDICAS A imagem médica digital é uma função f (x, y) em escala de níveis de cinza particionada em coordenadas espaciais que pode ser representada por uma matriz, cujos índices de linhas e de colunas identificam um ponto de imagem (pixel). O valor de cada pixel na matriz identifica o nível de cinza no ponto (x, y) (Figura 1), em uma escala de valores inteiros que representam preto (menor valor), branco (maior valor) e tons de cinza (valores intermediários). Imagens de TC e RM podem ser adquiridas de maneira volumétrica, em forma de um volume de fatias paralelas e uniformemente espaçadas, de maneira que um ponto da imagem representa um voxel, com “altura” igual à espessura de corte(15–17). É com essa matriz de valores numéricos representando a imagem que as ferramentas de processamento e análise trabalham(18). 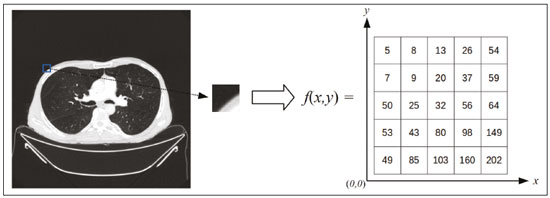 Figura 1. Representação da função (matriz) de uma imagem médica digital (corte axial de uma TC de tórax) em escala de cinza. No final da década de 1990 e início dos anos 2000, ocorreu uma grande revolução tecnológica na área da radiologia com a estruturação de ambientes radiológicos sem filme, totalmente digitalizados e integrados com outros sistemas de informação, dentro de um modelo denominado hospital digital(19). A base da radiologia sem filme é o sistema de arquivamento e comunicação de imagens (PACS, do inglês picture archiving and communication system), responsável por receber as imagens no padrão de comunicação de imagem digital em medicina (DICOM, do inglês digital imaging and communications in medicine) dos diversos aparelhos de aquisição (radiografia simples, ultrassonografia, TC e RM), deixá-las disponíveis para que especialistas ou outros sistemas computacionais possam acessá-las e ainda armazená-las de maneira organizada em grandes bases de dados(20). A integração do PACS com os sistemas de informação clínica HIS (do inglês hospital information system) e RIS (do inglês radiology information system) dos hospitais possibilitou também o desenvolvimento de diferentes modelos de auxílio computadorizado ao diagnóstico(21,22). A segmentação é uma das principais etapas no processamento e análise de imagens médicas, muitas vezes representando grandes desafios. Seu principal objetivo é dividir a imagem em partes que tenham correlação com a anatomia normal ou das lesões, ou seja, segmentar é separar anatomicamente tecidos e estruturas(23). Às vezes, a segmentação pode ser simplificada, utilizando formas definidas (circular, retangular) ou delineadas na imagem representando uma região de interesse (ROI, do inglês region of interest), que não necessariamente abrange todo um tecido, estrutura ou órgão. As técnicas para segmentação de imagens médicas são geralmente baseadas nas propriedades básicas de níveis de cinza, de descontinuidade (bordas) ou similaridade (após definir limiares ou usando algoritmo de crescimento de região – Figuras 2 e 3). Ainda mais, a segmentação pode ser manual, semiautomática (o usuário interfere em algum momento do processo) ou completamente automática (sem interferência do usuário)(24). 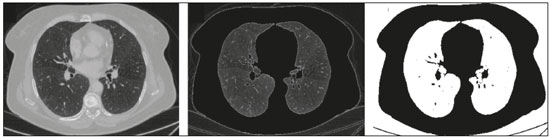 Figura 2. Segmentação semiautomática do pulmão em imagem de TC de tórax com 256 níveis de cinza e limiar de 115 unidades Hounsfield definido pelo usuário: a imagem de TC original (imagem à esquerda) é submetida a limiarização (detectar bordas) e posterior binarização (separação dos pulmões).  Figura 3. Segmentação semiautomática de um nódulo pulmonar neoplásico com crescimento de região em imagem de TC do tórax, após colocação da semente marcada pelo usuário (ponto ao centro do nódulo na primeira imagem à esquerda). O processo de extração de atributos de uma imagem consiste no cálculo de valores numéricos (descritores) que representem o seu conteúdo visual. Os atributos são obtidos por meio da execução de algoritmos, conhecidos como extratores de características. Esses algoritmos realizam procedimentos quantitativos nas imagens, como, por exemplo, construção de histogramas, classificação de texturas, reconhecimento de formas e contornos, estimativas de área e volume. Após a extração de características a partir desses algoritmos, os valores são armazenados em um vetor de atributos da imagem. Os atributos são basicamente classificados em três grupos principais: cor (níveis de cinza), textura e forma. A extração de atributos de níveis de cinza é a mais utilizada e pode ser realizada diretamente ou por análise do histograma da imagem. O histograma é a descrição da quantidade de níveis de cinza presentes na imagem, e o seu cálculo envolve apenas a contagem de pixels/voxels com as intensidades na escala de cinza (Figura 4)(3,7,25). Contudo, utilizar somente descritores de níveis de cinza ou histograma não fornece informações da distribuição espacial do conteúdo de uma imagem, esta podendo ser analisada pelos atributos de textura(24,26). Em algumas imagens médicas, regiões que possuem pixels/voxels similares são possíveis de se distinguir por causa das diferentes texturas (Figura 5). Atributos de textura se tornaram particularmente importantes em razão da sua capacidade de refletir os detalhes contidos dentro de uma lesão identificada na imagem médica(1,2). Os atributos de forma descrevem a borda da imagem e características geométricas extraídas do objeto segmentado, como contornos, junções, curvas e regiões poligonais(2,27). Caracterizar quantitativamente formas de objetos é uma tarefa complicada porque depende da eficiência dos algoritmos de segmentação. As lesões em imagens radiológicas do pulmão, por exemplo, muitas vezes apresentam opacidades ou estruturas adjacentes (por exemplo: vasos, mediastino – Figura 5), o que pode acarretar uma segmentação ruim e, consequentemente, pior caracterização por atributos de forma. 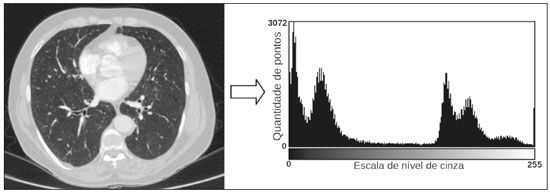 Figura 4. Exemplo de histograma de uma imagem axial de TC do tórax com 256 níveis de cinza. O histograma mostra a distribuição dos pixels ou voxels de acordo com os níveis de cinza (ou unidades Hounsfield, se necessário). 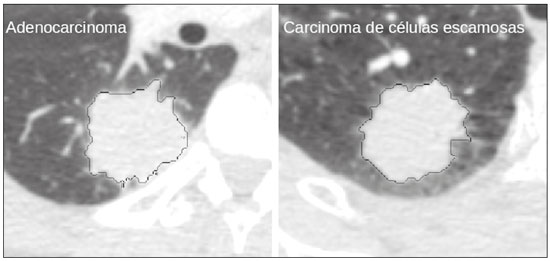 Figura 5. Segmentação de nódulos pulmonares na TC de tórax em dois pacientes diferentes, ambos com contornos irregulares e apresentando relação com vasos adjacentes ou a parede torácica, fatores que dificultam a segmentação adequada das bordas das lesões. Nesses casos, a análise da textura pode auxiliar na segmentação correta. A seleção de atributos relevantes é outra etapa importante, em razão da grande gama de atributos que podem ser extraídos da imagem. Muitas vezes as características podem ser irrelevantes para uma determinada análise, ou os dados podem apresentar redundância, inserindo ruído ou inconsistências ao vetor de atributos(28). Dessa forma, é necessário realizar uma seleção das características mais relevantes de acordo com a classe de investigação clínica (diagnóstico ou desfecho). Diversos algoritmos foram criados com o objetivo de reduzir a dimensionalidade espacial do vetor, podendo ser classificados em três tipos principais: filtro, wrapper e embarcado. Boa parte dos algoritmos utiliza recurso de aprendizado de máquina e alguns realizam a seleção dos atributos utilizando redes neurais artificiais (RNAs), árvores de decisão e florestas aleatórias(11). CLASSIFICAÇÃO DAS IMAGENS, APRENDIZADO DE MÁQUINA E APRENDIZADO PROFUNDO Classificar a imagem significa, em geral, defini-la dentro de uma categoria pré-estabelecida, como normal versus patológico. Uma das áreas mais estudadas em inteligência artificial e na classificação de imagens médicas é a de aprendizado de máquina (do inglês machine learning). Esta técnica permite a identificação de padrões com base em casos e experimentos anteriores, assim como ocorre com a inteligência humana(29). Métodos de aprendizado de máquina têm sido aplicados para classificação de diferentes modalidades de imagens médicas, utilizando uma variedade de atributos, para diversas doenças e ferramentas, como o CAD (do inglês computer aided diagnosis/detection) e a radiômica(25,29,30). A criação de um método de aprendizado de máquina envolve a criação de uma função de treinamento para um conjunto de dados (os vetores de atributos, no caso de classificação de imagens médicas), fazendo-se uso de um mecanismo de inferência lógica. Quando as classes (diagnósticos ou desfechos clínicos) para a decisão final do modelo são pré-estabelecidas, o processo de treinamento é supervisionado. Quando não existe classe definida, o processo é não supervisionado. Neste último caso, o algoritmo visa à formação de agrupamentos de amostras semelhantes (“exames com padrão semelhante”), podendo ou não estar relacionado a uma condição conhecida (por exemplo: uma doença)(31,32). As RNAs são um dos mais tradicionais métodos de aprendizado de máquina e são bastante utilizadas em tarefas de classificação de imagens médicas(25,29,30). Essas redes neurais foram propostas tendo como referência as estruturas do sistema nervoso central humano(33), com modelos matemáticos descritos em forma de camadas, sendo cada camada composta por um conjunto de N neurônios. O tipo de RNA mais conhecido é o perceptron de multicamadas (MLP, do inglês multilayer perceptron). Tradicionalmente, a rede tipo MLP possui uma camada de entrada (cujos neurônios correspondem aos atributos da imagem), uma camada de saída (cujos neurônios correspondem às classes/desfechos) e um conjunto de camadas ocultas intermediárias (cujos neurônios correspondem aos pontos de ajuste das funções de ativação) (Figura 6). 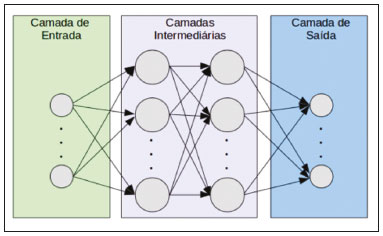 Figura 6. Arquitetura de uma RNA de multicamadas. A camada de entrada recebe as informações dos atributos. A camada de saída representa as classes ou desfechos (por exemplo: normal versus patológico). Uma das áreas de inteligência artificial mais recentes que vem ganhando atenção da comunidade científica é a de aprendizado profundo (do inglês deep learning)(16). Métodos tradicionais de aprendizado de máquina possuem limitações no processamento de dados, principalmente relacionados à necessidade de segmentação e desenvolvimento de extratores de atributos para representar as imagens e servir de entrada para os classificadores(34). Diante disso, pesquisadores passaram a desenvolver algoritmos que integrassem os processos de extração de atributos e classificação de imagens dentro da própria rede neural. Dessa maneira, na técnica de aprendizado profundo, é minimizada a necessidade de pré-processamento ou segmentação. O método, porém, também apresenta desvantagens, como a necessidade de um conjunto muito grande de imagens (centenas a milhares), maior dependência da qualidade dos exames e dados clínicos e a dificuldade de se identificar a lógica utilizada (black box do processamento). O método de aprendizado profundo que mais ganhou notoriedade em medicina é a rede neural convolucional (RNC, do inglês convolutional neural network). Uma RNC é composta basicamente por três tipos de camadas: a primeira camada (convolucional) detecta e extrai os atributos; a segunda camada (pooling) seleciona e reduz a quantidade de atributos; e a terceira camada (totalmente conectada) serve para integrar todas as características extraídas pelas camadas anteriores, normalmente utilizando uma rede neural do tipo MLP, para realizar a classificação final da imagem, dada pela predição da classe com maior probabilidade (Figura 7)(35). 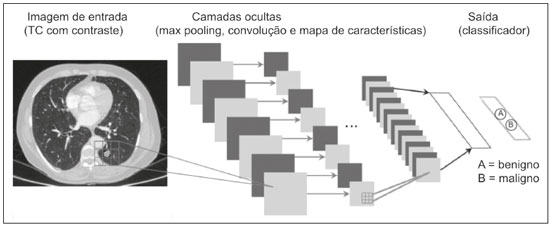 Figura 7. Imagem de TC de tórax com nódulo pulmonar como entrada em uma rede neural convolucional para análise utilizando técnica de aprendizado profundo. Outra etapa importante do processo de aprendizado de máquina é a validação e avaliação de performance. Dado um conjunto de imagens, um classificador de aprendizado de máquina deve utilizar no mínimo dois subconjuntos diferentes para realizar o treinamento do algoritmo e a validação do modelo preditivo. Em radiologia, uma estratégia muito utilizada é a validação cruzada. Nela, as amostras são separadas em N subconjuntos, sendo N − 1 para treino e 1 para teste(13,36). Outra estratégia, que diminui o risco de sobreajuste do modelo, é baseada em três subconjuntos de amostras: um para treino, um para validação e um conjunto independente somente para teste(29,37). A avaliação da performance é geralmente baseada no cálculo da acurácia, sensibilidade, especificidade e área sob a curva ROC (do inglês receiver operating characteristic). O valor da AUC (do inglês area under the ROC curve) corresponde à área da região sob a curva ROC, e quanto mais próximo de 1 (escala de 0 a 1), maior a acurácia do método (Figura 8). 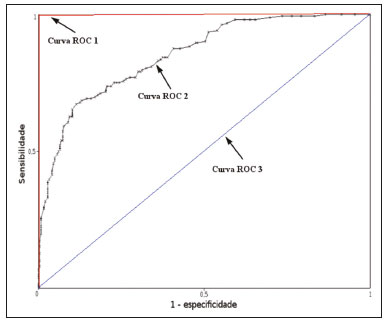 Figura 8. Exemplo de curvas ROC: a curva 1 representa um teste com performance perfeita de área igual a 1; a curva 2 representa um teste com performance intermediária de área igual a 0,75; e a curva 3 é um teste aleatório com área igual a 0,5. Essas ferramentas que utilizam inteligência artificial, aprendizado de máquina e aprendizado profundo podem ser empregadas de diferentes maneiras na análise de imagens médicas. Na área de radiologia e diagnóstico por imagem, elas têm sido aplicadas principalmente no diagnóstico/detecção auxiliado por computador, na recuperação de imagens baseada em conteúdo (CBIR, do inglês content based imaging retrieval) e na radiômica/radiogenômica. DIAGNÓSTICO/DETECÇÃO AUXILIADO POR COMPUTADOR Ferramentas de CAD foram inicialmente desenvolvidas para auxiliar na interpretação dos achados radiológicos e na identificação precoce de doenças, especialmente tumores de mama e pulmão(7,10). O objetivo do CAD é melhorar a acurácia e consistência do diagnóstico por imagem, mediante o uso da sugestão de resposta (“segunda opinião”) fornecida principalmente por técnicas de processamento de imagens, visão computacional e aprendizado de máquina(1,3,38). Historicamente, os primeiros sistemas CAD surgiram no final da década de 1980 e eram baseados no processamento de filmes radiológicos digitalizados. Eles foram fundamentalmente concebidos para trabalhar como uma segunda leitura do exame em programas de rastreamento populacional de câncer (ajudar a detectar nódulos e microcalcificações em mamografias). Posteriormente, sistemas semelhantes foram utilizados para detectar e classificar nódulos pulmonares em exames de radiografia simples e TC de tórax. Mais recentemente, esses sistemas têm sido utilizados também para auxiliar no diagnóstico de doença de Alzheimer em exames de medicina nuclear. Nesses modelos tradicionais de CAD, a ideia é que a segunda leitura seja feita pelo computador em lugar de um segundo médico radiologista (Figura 9)(25,29). 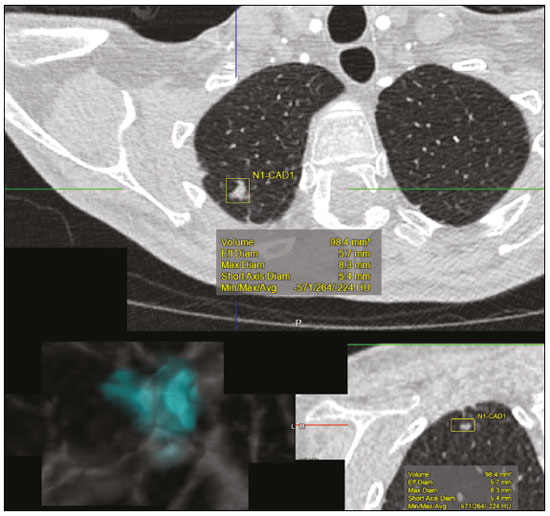 Figura 9. Exemplo de ferramenta de CAD para detecção e análise de nódulos pulmonares. O programa não somente indica a presença do nódulo pulmonar apical direito, como fornece informações quantitativas e tridimensionais desse nódulo. Com o desenvolvimento da inteligência artificial e das novas ferramentas de aprendizado de máquina, os sistemas de auxílio diagnóstico se expandiram sobremaneira e passaram a ser utilizados nas mais diferentes tarefas, com todo tipo de imagem médica. Citamos como exemplo o número surpreendente de apresentações (aulas, trabalhos, sessões) no último congresso da Sociedade Americana de Radiologia (RSNA meeting 2018), atualmente o maior congresso da especialidade no mundo em número de participantes e trabalhos. Foram 237 registros de eventos contendo um dos tópicos relacionados: inteligência artificial, aprendizado de máquina ou aprendizado profundo (https://rsna2018.rsna.org/program/index.cfm). Alguns exemplos incluem: detecção automática de nódulos pulmonares, pneumonia, pneumotórax e derrame pleural na radiografia simples de tórax; detecção e quantificação de enfisema, estimativa do risco de malignidade de nódulo pulmonar, chance de invasão local do câncer pulmonar e risco cardiovascular na TC de tórax; análise automática da função cardíaca em RM do coração; e muitos outros. Dessa maneira, as possibilidades de desenvolvimento de ferramentas de auxílio diagnóstico computadorizado tornaram-se quase que ilimitadas(39,40). RECUPERAÇÃO DE IMAGENS BASEADA EM CONTEÚDO O conceito de CBIR refere-se à busca por imagens cujos conteúdos são similares a um caso de referência utilizando informações derivadas das próprias imagens, representadas por seu conteúdo intrínseco (vetor de atributos), em vez de textos associados (laudos) ou anotações externas(41). A CBIR tem sido descrita como uma das ferramentas computacionais mais promissoras, pois ela tem potencial de aplicação clínica, no ensino e na pesquisa(42,43). Esta ferramenta pode ser muito útil no ambiente clínico do dia-a-dia, pois pode auxiliar o radiologista na interpretação diagnóstica dos exames, ou aumentar seu grau de confiança, valendo-se de um modelo de decisão baseada em exemplos semelhantes(22). Atualmente, o mais comum é que o médico, diante de uma dúvida diagnóstica, procure na internet em sítios de busca ou sítios especializados em radiologia, por textos ou casos semelhantes, utilizando palavras-chave ou se valendo de sua suspeita diagnóstica (por exemplo: buscar no Google por “nódulo pulmonar na TC de tórax” ou “hamartoma pulmonar”). A CBIR substitui esse tipo de tarefa de maneira mais eficaz, trazendo ao médico casos semelhantes à imagem para a qual ele busca definição. Assim, enquanto os sistemas CAD executam tarefas de classificação de imagens, fornecendo em geral uma única resposta (tem ou não tem lesão, é benigno ou maligno), os sistemas CBIR executam buscas de imagens médicas por similaridade, fornecendo um conjunto de casos similares a um caso desconhecido indicado pelo médico. RADIÔMICA E RADIOGENÔMICA O sufixo -ômica é um termo originado na biologia molecular para descrever a caracterização detalhada de moléculas como o DNA (genômica) e as proteínas (proteômica). A radiômica tem sido descrita como uma extensão do CAD que associa as características quantitativas (atributos) das imagens médicas com dados e desfechos clínicos do paciente, permitindo não somente o diagnóstico, mas também fornecendo informações prognósticas e de resposta terapêutica(7,25). Em face dos recentes avanços nas terapias-alvo e imunoterapias, principalmente no tratamento das neoplasias, a necessidade de uma abordagem de análise robusta de imagens médicas se tornou imperativa, e a radiômica tem potencial para prover isto de maneira não invasiva, rápida e de baixo custo(12). A análise radiômica define um processo de extração massiva de atributos das imagens médicas (dezenas a centenas), de inserção desses atributos em bases de dados com informações clínicas do paciente, que permitam seu compartilhamento e sua análise(44). O volume de dados em saúde vem crescendo em um ritmo acelerado nos últimos anos, caracterizando o que alguns autores chamam de “era do big data” da saúde, e esses dados eletrônicos estão disponíveis em volumosas quantidades nos sistemas de informação dos grandes hospitais e centros de saúde(45). Diante da grande quantidade de atributos e das inúmeras possibilidades de processamento, alguns autores passaram a desenvolver e sugerir o uso de uma assinatura radiômica, em que são escolhidos os atributos mais significativos, de diferentes categorias, analisados e testados com protocolos acurados e algoritmos reprodutíveis(46). Já há na literatura médica muitos trabalhos usando a radiômica em diferentes tipos de exames de imagem para estudo de diferentes doenças, a maioria aplicada à oncologia, como, por exemplo, no estudo das neoplasias de pulmão e rim em imagens de TC e PET/TC, neoplasias de próstata, mama, glioma cerebral e carcinoma hepatocelular em imagens de RM(7,14,36). Quando o foco da radiômica é o estudo da correlação entre os padrões radiológicos e genômicos (conjunto de genes), o processo passa a receber o nome de radiogenômica(47). Estudos têm demonstrado que atributos das imagens médicas e padrões de expressão e mutações genéticas possuem associações significativas, mostrando que a análise radiogenômica pode demonstrar diferentes mecanismos biológicos por meio de dispositivos matemáticos e computacionais, possibilitando a decodificação de fenótipos das doenças por meio de métodos não invasivos(48,49). A radiogenômica vem também sendo empregada em estudos que analisam a heterogeneidade intratumoral, que é a presença de múltiplas sub-regiões teciduais e genéticas em um mesmo tumor, relacionada à recidiva da doença e à resistência terapêutica. A radiogenômica é capaz de quantificar a complexidade espacial do tumor e identificar essas sub-regiões fenotípicas/genotípicas (Figura 10)(50). 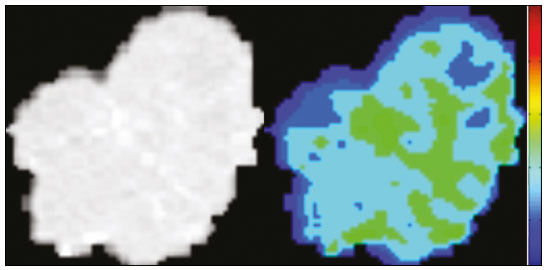 Figura 10. Quantificação da heterogeneidade intratumoral de um adenocarcinoma pulmonar em imagem de TC do tórax, para avaliação radiômica/radiogenômica. A escala de cores se refere a um atributo extraído da imagem refletindo as sub-regiões teciduais e genéticas do tumor. CONCLUSÃO A radiologia vem passando por importantes atualizações com a revolução tecnológica que acontece no mundo: primeiro, com a digitalização dos ambientes radiológicos; depois, com a evolução das técnicas de visão computacional e inteligência artificial no desenvolvimento de sistemas de auxílio diagnóstico; e mais recentemente, com o amadurecimento dos modelos computacionais de suporte a decisões clínicas e predição prognóstica. Neste trabalho apresentamos e discutimos os principais conceitos relacionados à análise computadorizada das imagens, incluindo aspectos de inteligência artificial aplicada à medicina de precisão. Os autores acreditam que essas ferramentas irão mudar o jeito de trabalhar dos médicos radiologistas e demais especialistas que trabalham com exames de imagem, e provavelmente, em um futuro muito próximo, mudar a perspectiva de trabalho de todos os envolvidos na área da saúde. Entretanto, é temerosa a afirmação de que o médico radiologista e demais especialistas sejam completamente substituídos pelo computador. Talvez, tarefas e exames simples (por exemplo: avaliação de escoliose ou idade óssea na radiografia simples) possam até ser completamente realizados e interpretados pela máquina, porém, o papel do médico na verificação/validação do resultado, correlação clinicoepidemiológica e decisão terapêutica não deverá ser ameaçado. Além, claro, das questões éticas e legais relacionadas à responsabilidade sobre o exame médico. A inteligência artificial certamente vai ajudar a “baixar a pilha” de exames, reduzir o tempo de ação nos casos urgentes, agilizar a interpretação e emissão dos relatórios, aumentar o grau de confiança nos diagnósticos, tornar mais objetiva e reprodutível a análise das imagens, oferecer informações prognósticas de maneira mais fidedigna, auxiliar no ensino e aprendizado da imaginologia, e, por fim, inserir definitivamente a radiologia no conceito de medicina de precisão e avaliação multidisciplinar do paciente. Na prática, imagina-se que a primeira mudança será que os médicos radiologistas de hoje, que em sua maioria trabalham utilizando uma estação de trabalho com duas telas de computador (uma com a ferramenta de visualização das imagens, outra com o sistema para emissão do relatório e acesso aos dados clínicos e radiológicos), provavelmente passarão a trabalhar com três telas, e na terceira estará disponível a análise da inteligência artificial. Mais importante que temer, é preciso que o médico radiologista se prepare, aprenda e se adapte, pois as mudanças são inevitáveis. REFERÊNCIAS 1. Azevedo-Marques PM, Mencattini A, Salmeri R, et al. Medical image analysis and informatics. Boca Raton, FL: CRC Press; 2017. 2. Akgül CB, Rubin DL, Napel S, et al. Content-based image retrieval in radiology: current status and future directions. J Digit Imaging. 2011;24:208–22. 3. Doi K. Computer-aided diagnosis in medical imaging: historical review, current status and future potential. Comput Med Imaging Graph. 2007;31:198–211. 4. Koenigkam-Santos M, Paula DW, Gompelmann D, et al. Endobronchial valves in severe emphysematous patients: CT evaluation of lung fissures completeness, treatment radiological response and quantitative emphysema analysis. Radiol Bras. 2013;46:15–22. 5. Koenigkam-Santos M, Cruvinel DL, Menezes MB, et al. Quantitative computed tomography analysis of the airways in patients with cystic fibrosis using automated software: correlation with spirometry in the evaluation of severity. Radiol Bras. 2016;49:351–7. 6. Wada DT, Pádua AI, Lima Filho MO, et al. Use of computed tomography and automated software for quantitative analysis of the vasculature of patients with pulmonary hypertension. Radiol Bras. 2017;50:351–8. 7. Gillies RJ, Kinahan PE, Hricak H. Radiomics: images are more than pictures, they are data. Radiology. 2016;278:563–77. 8. Koenigkam Santos M, Muley T, Warth A, et al. Morphological computed tomography features of surgically resectable pulmonary squamous cell carcinomas: impact on prognosis and comparison with adenocarcinomas. Eur J Radiol. 2014;83:1275–81. 9. Cooper WA, O’Toole S, Boyer M, et al. What’s new in non-small cell lung cancer for pathologists: the importance of accurate subtyping, EGFR mutations and ALK rearrangements. Pathology. 2011;43:103–15. 10. Aerts HJWL. The potential of radiomic-based phenotyping in precision medicine: a review. JAMA Oncol. 2016;2:1636–42. 11. Constanzo J, Wei L, Tseng HH, et al. Radiomics in precision medicine for lung cancer. Transl Lung Cancer Res. 2017;6:635–47. 12. Giardino A, Gupta S, Olson E, et al. Role of imaging in the era of precision medicine. Acad Radiol. 2017;24:639–49. 13. Aerts HJWL, Grossmann P, Tan Y, et al. Defining a radiomic response phenotype: a pilot study using targeted therapy in NSCLC. Sci Rep. 2016;6:33860. 14. Keek SA, Leijenaar RT, Jochems A, et al. A review on radiomics and the future of theranostics for patient selection in precision medicine. Br J Radiol. 2018;91:20170926. 15. MacMahon H, Naidich DP, Goo JM, et al. Guidelines for management of incidental pulmonary nodules detected on CT images: from the Fleischner Society 2017. Radiology. 2017;284:228–43. 16. Bartholmai BJ, Koo CW, Johnson GB, et al. Pulmonary nodule characterization, including computer analysis and quantitative features. J Thorac Imaging. 2015;30:139–56. 17. Truong MT, Ko JP, Rossi SE, et al. Update in the evaluation of the solitary pulmonary nodule. Radiographics. 2014;34:1658–79. 18. Bushberg JT, Seibert JA, Leidholdt EM Jr, et al. The essential physics of medical imaging. 3rd ed. Philadelphia, PA: Lippincott Williams & Wilkins; 2012. 19. Pavlopoulos SA, Delopoulos AN. Designing and implementing the transition to a fully digital hospital. IEEE Trans Inf Technol Biomed. 1999;3:6–19. 20. Ponciano-Silva M, Bugatti PH, Reis RM, et al. A differential method for representing spinal MRI for perceptual-CBIR. In: Ruiz-Shulcloper J, Sanniti di Baja, editors. Progress in pattern recognition, image analysis, computer vision, and applications. CIARP 2013. Berlin, Heidelberg: Springer-Verlag; 2013. p. 464–71. 21. Traina AJM, Bedo MVN, Santos LFD, et al. Making content-based medical image retrieval systems worth for computer-aided diagnosis: from theory to application. In: Azevedo-Marques PM, Mencattini A, Salmeri M, et al., editors. Medical image analysis and informatics computer-aided diagnosis and therapy. Boca Raton, FL: CRC Press; 2017. p. 24. 22. Ponciano-Silva M, Souza JP, Bugatti PH, et al. Does a CBIR system really impact decisions of physicians in a clinical environment? In: Proceedings of the 26th IEEE International Symposium on Computer- Based Medical Systems. IEEE; 2013. p. 41–6. 23. Sonka M, Hlavac V, Boyle R. Image processing, analysis and machine vision. Boston, MA: Springer; 1993. 24. Gonzalez RC, Woods RE. Digital image processing. Upper Saddle River, NJ: Prentice Hall; 2007. 25. Giger ML. Machine learning in medical imaging. J Am Coll Radiol. 2018;15(3 Pt B):512–20. 26. Parker JR. Algorithms for image processing and computer vision. 2nd ed. Indianapolis, IN: Wiley; 2011. 27. Echegaray S, Gevaert O, Shah R, et al. Core samples for radiomics features that are insensitive to tumor segmentation: method and pilot study using CT images of hepatocellular carcinoma. J Med Imaging (Bellingham). 2015;2:041011. 28. Frighetto-Pereira L, Rangayyan RM, Metzner GA, et al. Shape, texture and statistical features for classification of benign and malignant vertebral compression fractures in magnetic resonance images. Comput Biol Med. 2016;73:147–56. 29. Erickson BJ, Korfiatis P, Akkus Z, et al. Machine learning for medical imaging. Radiographics. 2017;37:505–15. 30. Choy G, Khalilzadeh O, Michalski M, et al. Current applications and future impact of machine learning in radiology. Radiology. 2018;288:318–28. 31. Leger S, Zwanenburg A, Pilz K, et al. A comparative study of machine learning methods for time-to-event survival data for radiomics risk modelling. Sci Rep. 2017;7:13206. 32. Parmar C, Grossmann P, Bussink J, et al. Machine learning methods for quantitative radiomic biomarkers. Sci Rep. 2015;5:13087. 33. Haykin S. Neural networks: a comprehensive foundation. Upper Saddle River, NJ: Prentice Hall; 1994. 34. LeCun Y, Bengio Y, Hinton G. Deep learning. Nature. 2015;521: 436–44. 35. Soffer S, Ben-Cohen A, Shimon O, et al. Convolutional neural networks for radiologic images: a radiologist’s guide. Radiology. 2019;290: 590–606. 36. Lambin P, Leijenaar RTH, Deist TM, et al. Radiomics: the bridge between medical imaging and personalized medicine. Nat Rev Clin Oncol. 2017;14:749–62. 37. Lee JG, Jun S, Cho YW, et al. Deep learning in medical imaging: general overview. Korean J Radiol. 2017;18:570–84. 38. Ferreira JR Jr, Azevedo-Marques PM, Oliveira MC. Selecting relevant 3D image features of margin sharpness and texture for lung nodule retrieval. Int J Comput Assist Radiol Surg. 2017;12:509–17. 39. Nam JG, Park S, Hwang EJ, et al. Development and validation of deep learning-based automatic detection algorithm for malignant pulmonary nodules on chest radiographs. Radiology. 2019;290:218–28. 40. Tao Q, Yan W, Wang Y, et al. Deep learning-based method for fully automatic quantification of left ventricle function from cine MR images: a multivendor, multicenter study. Radiology. 2019;290:81–8. 41. Souza JP. Modelo de qualidade para o desenvolvimento e avaliação da viabilidade clínica de sistemas de recuperação de imagens médicas baseadas em conteúdo (Tese). São Carlos, SP: Universidade de São Paulo; 2012. [cited 2019 April 1]. Available from: www.teses.usp.br/teses/disponiveis/82/82131/tde-15022013-142541/pt-br.psp. 42. Ferreira Junior JR, Oliveira MC. Evaluating margin sharpness analysis on similar pulmonary nodule retrieval. In: 2015 IEEE 28th International Symposium on Computer-Based Medical Systems. IEEE; 2015. p. 60–5. 43. Bugatti PH, Kaster DS, Ponciano-Silva M, et al. PRoSPer: perceptual similarity queries in medical CBIR systems through user profiles. Comput Biol Med. 2014;45:8–19. 44. Larue RTHM, Defraene G, De Ruysscher D, et al. Quantitative radiomics studies for tissue characterization: a review of technology and methodological procedures. Br J Radiol. 2017;90:20160665. 45. Filonenko E, Seeram E. Big data: the next era of informatics and data science in medical imaging – a literature review. J Clin Exp Radiol. 2018;1(1). 46. Coroller TP, Grossmann P, Hou Y, et al. CT-based radiomic signature predicts distant metastasis in lung adenocarcinoma. Radiother Oncol. 2015;114:345–50. 47. Thawani R, McLane M, Beig N, et al. Radiomics and radiogenomics in lung cancer: a review for the clinician. Lung Cancer. 2018;115:34–41. 48. Gevaert O, Echegaray S, Khuong A, et al. Predictive radiogenomics modeling of EGFR mutation status in lung cancer. Sci Rep. 2017;7:41674. 49. Aerts HJWL, Velazquez ER, Leijenaar RTH, et al. Decoding tumour phenotype by noninvasive imaging using a quantitative radiomics approach. Nat Commun. 2014;5:4006. 50. Lee G, Lee HY, Park H, et al. Radiomics and its emerging role in lung cancer research, imaging biomarkers and clinical management: state of the art. Eur J Radiol. 2017;86:297–307. 1. Centro de Ciências das Imagens e Física Médica (CCIFM) da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP-USP), Ribeirão Preto, SP, Brasil 2. Escola de Engenharia de São Carlos da Universidade de São Paulo (EESC-USP), São Carlos, SP, Brasil 3. Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP-USP), Ribeirão Preto, SP, Brasil a. https://orcid.org/0000-0002-7160-4691 b. https://orcid.org/0000-0002-8202-588X c. https://orcid.org/0000-0002-6433-4849 d. https://orcid.org/0000-0002-3908-9538 e. https://orcid.org/0000-0002-7436-5315 f. https://orcid.org/0000-0002-7271-2774 Correspondência: Dr. Marcel Koenigkam Santos Avenida Bandeirantes, 3900, Campus Universitário, Monte Alegre Ribeirão Preto, SP, Brasil, 14048-900 E-mail: marcelk46@fmrp.usp.br Recebido para publicação em 29/3/2019 Aceito, após revisão, em 15/4/2019 |
|
Av. Paulista, 37 - 7° andar - Conj. 71 - CEP 01311-902 - São Paulo - SP - Brazil - Phone: (11) 3372-4544 - Fax: (11) 3372-4554