Radiologia Brasileira - Publicação Científica Oficial do Colégio Brasileiro de Radiologia
AMB - Associação Médica Brasileira CNA - Comissão Nacional de Acreditação
 Vol. 40 nº 4 - Jul. / Ago. of 2007
Vol. 40 nº 4 - Jul. / Ago. of 2007
|
ARTIGO ORIGINAL
|
|
|
|
|
Autho(rs): Marcelo Costa Oliveira, Paulo Mazzoncini de Azevedo-Marques, Walfredo da Costa Cirne Filho |
|
|
Descritores: Recuperação de imagens baseada em conteúdo, Análise de textura, Registro de imagens, Grades computacionais |
|
|
Resumo:
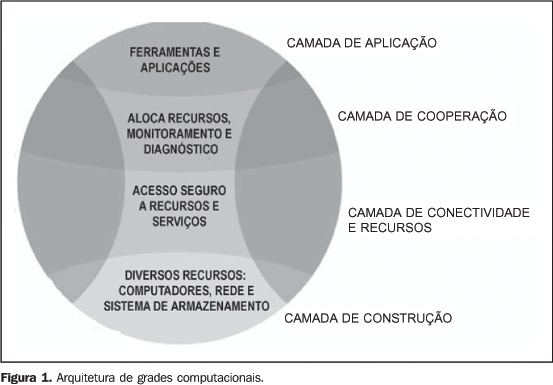
INTRODUÇÃO O volume de dados produzidos nos hospitais e centros médicos está em rápida ascensão. A produção anual de dados gerados por exames de imagens nos grandes centros de radiologia é da ordem de 2 terabytes. Isto é devido à obtenção e armazenamento de dados dos pacientes, resultantes da crescente importância e utilização dos exames de imagem. É enorme a quantidade de dados que necessitam ser armazenados e indexados de forma inteligente e segura, pois são peças fundamentais no diagnóstico clínico(1,2). O crescente uso de programas de computador para auxílio diagnóstico (computer-aided diagnosis – CAD) está relacionado ao rápido desenvolvimento de algoritmos computacionais aplicados à medicina. O objetivo do CAD é melhorar a acurácia diagnóstica, assim como aprimorar a consistência na interpretação de imagens diagnósticas, mediante o uso da sugestão de resposta diagnóstica fornecida por algum computador(3). Porém, algumas ferramentas CAD que apresentam ótimos resultados ainda não são utilizadas na rotina clínica por apresentarem alto custo computacional, limitando seu uso a centros possuidores de computadores de alta capacidade(4). As dificuldades em aplicar estes algoritmos CAD na rotina clínica e as limitações ainda existentes para o armazenamento, processamento, busca e recuperação de imagens em grandes bases de dados vem motivando empresas e instituições de pesquisas a encontrarem novas soluções para essas tarefas(5,6). Grade computacional A tecnologia de grade computacional (GC) representa a mais recente e promissora ferramenta na área de computação distribuída. De maneira sucinta, a computação distribuída é uma coleção de computadores independentes que se apresenta ao usuário como um sistema único e consistente. GC permitem a integração de computadores que estão amplamente distribuídos e conectados por meio de redes de longas distâncias. Com esta capacidade de integração de computadores, cria-se um sistema de computação virtual ou cooperativo para solucionar problemas referentes ao armazenamento e acesso de dados em massa e ao processamento de aplicações com alto custo computacional(6,7). A tecnologia de GC oferece um ambiente único para compartilhar, armazenar e processar dados. Ainda, permite à comunidade médica utilizar uma única base de dados distribuída, capaz de prover o compartilhamento de recursos, dados e conhecimento. Todos estes recursos permitem maior interação entre os centros médicos, inclusive oferecendo novas oportunidades a pequenas clínicas e laboratórios de pesquisa com poucos recursos computacionais(7). Além disso, a GC oferece uma infra-estrutura flexível, escalonável, menos sujeita a catástrofes e, portanto, mais confiável, sendo capaz de garantir acesso seguro a qualquer aplicação que a utilize. Diferentemente da computação distribuída e da metodologia de grupos de terminais ou de estações de trabalho ligadas a uma unidade de controle servidora ou grupo de unidades servidoras (clusters technology) computacionais, os recursos de GC possuem autonomia administrativa e heterogeneidade de sistema. Estas duas características permitem melhor escalonamento e robustez às aplicações. Entretanto, as mesmas características exigem que os componentes da GC devam seguir padrões visando ao escalonamento e compartilhamento de recursos computacionais de forma aberta(8). Foster e Kesselman(9) apresentam proposta de arquitetura e de componentes de uma GC. A arquitetura formal é composta por quatro camadas (Figura 1). A camada de construção é o nível mais baixo e representa os recursos físicos e dispositivos que os usuários da GC desejam compartilhar e acessar (computadores, rede, sistema de arquivos, catálogos, programas computacionais e instrumentos digitais). Logo acima da camada de construção está a camada de recursos e conectividade, responsável pela comunicação e autenticação necessárias para a troca de recursos, validação do usuário, monitoramento e controle sobre as operações de compartilhamento de recursos. A terceira camada ou camada de cooperação possui os protocolos e realiza os serviços responsáveis pelas transações de recursos (descoberta e alocação de recursos, monitoramento e diagnóstico da funcionalidade dos serviços, replicação de dados e políticas que regulamentam privilégios de usuários no acesso aos recursos da GC). A camada de aplicação do usuário é o mais alto nível e é responsável por invocar todas as outras camadas.
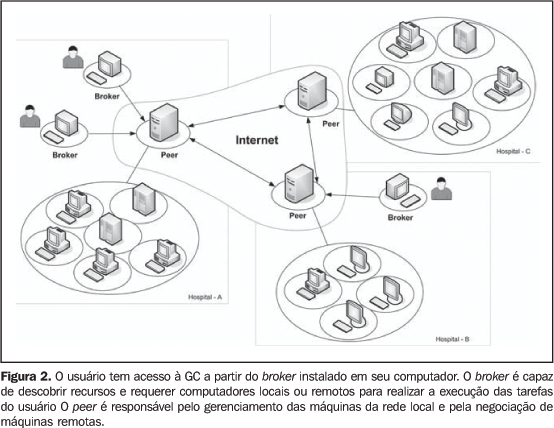
Existe grande número de projetos relacionados à GC descritos na literatura (por exemplo: Globus(10), Legion(11), Condor(12) e OurGrid(13)) que utilizam diversas tecnologias e são em grande maioria direcionados a determinadas áreas e propósitos, como aplicações de processamento e armazenamento de dados, portais Web e serviços de infra-estutura para colaboração entre instituições(14,15) . A utilização básica da GC pelo usuário é realizada por meio de uma interface de software que permite ao computador do usuário se comunicar com o centro de processamento da GC, conhecido como broker. Este é capaz de encontrar os recursos necessários à execução das tarefas. Ao término da execução da tarefa, o broker retorna o resultado da aplicação ao usuário(16). A Figura 2 ilustra o funcionamento básico do projeto OurGrid.
Recuperação de imagens médicas baseada em conteúdo Dentre as várias técnicas de CAD, os sistemas de recuperação de imagens médicas baseada em conteúdo (RIBC) são os grandes beneficiados da tecnologia de GC devido às suas características e necessidades: intensidade e complexidade de processamento e grande volume de imagens armazenadas(17). Por intermédio da RIBC é possível, a partir de uma imagem de referência, encontrar imagens similares contidas em uma ou diversas bases de imagens utilizando atributos inerentes das imagens. No processo de decisões clínicas, a RIBC oferece grandes benefícios, sendo capaz de recuperar imagens de mesma modalidade, região anatômica e alterações estruturais provocadas por determinadas doenças. Assim, a RIBC despertou rápido interesse na comunidade médica, em virtude da sua capacidade de recuperar imagens já diagnosticadas para comparar com uma imagem em estudo, permitindo ao especialista confirmar a sua hipótese diagnóstica(18). Embora parte dessa informação possa estar contida no cabeçalho da imagem médica, esta rotulação textual pode apresentar alta taxa de erro, tendo sido relatados casos de até 16%(19). É amplamente enfatizada nos trabalhos científicos a necessidade de adotar métodos de acesso alternativo aos dados cadastros manualmente inseridos no cabeçalho das imagens médicas(20–23). Além das técnicas de suporte à decisão clínica, pesquisa e ensino também são grandes beneficiários dos sistemas RIBC. No ensino, a RIBC auxilia os docentes e discentes na utilização de repositórios de imagens educacionais e na análise visual dos resultados encontrados. Além dos estudos baseados no diagnóstico e região anatômica, a análise de casos visualmente similares, porém com diferentes diagnósticos, resulta no aprimoramento da qualidade educacional(24). RIBC é uma das técnicas de visão computacional mais estudadas nos últimos dez anos e baseia-se nas características visuais da imagem, que são agrupadas em três classes: cor, textura e forma(25). Estes atributos possibilitam o desenvolvimento de ferramentas computacionais robustas e capazes de caracterizar imagens pelo seu próprio conteúdo, adicionando vantagens à identificação de imagens baseadas somente em descritores textuais que formam a classificação tradicional de arquivos de imagens médicas(23). A distribuição dos níveis de cinza é a característica mais simples de ser caracterizada. Sua caracterização é realizada pela comparação entre histogramas de níveis de cinza utilizando o somatório de diferenças absolutas ou quadráticas sobre o número de elementos da imagem (pixels) de cada intensidade de cinza. Por apresentarem ambigüidade (em que diferentes imagens podem gerar o mesmo somatório), não são eficazes para o todo da RIBC, mas por sua simplicidade e baixo custo computacional podem e devem ser utilizadas como filtros primários para outros métodos mais elaborados e de alto custo. Características baseadas em textura buscam quantificar a variação e a escala de intensidade da imagem. Um dos métodos mais utilizados na literatura para extrair atributos de textura é a matriz de co-ocorrência(24). Haralick et al.(26) definiram os atributos de textura que podem ser obtidos a partir da matriz de co-ocorrência com propósitos de discriminação de textura. Aproximadamente 20 funções estatísticas são propostas pela literatura para obtenção de informação a partir da matriz de co-ocorrência(27). Algumas das funções mais importantes e que produzem satisfatória classificação das texturas são entropia, inércia, energia, matiz, momento da diferença inverso, proeminência, correlação e variância(27–33). A recuperação de imagens baseada em forma é um dos problemas mais difíceis de serem tratados pelos sistemas de RIBC. Isto é devido à complexidade em realizar segmentação automática de imagens médicas. Após serem segmentadas, as estruturas são descritas pelas características de forma, incluindo informações sobre rotação, translação e escala(34). Outra técnica de RIBC descrita na literatura é o registro de imagens(5,17). Esta técnica calcula uma transformação rígida bidimensional de coordenadas englobando rotação, translação e escala, buscando o máximo "casamento" (matching) entre duas imagens ou entre dois volumes de imagens. A transformação rígida é baseada na minimização do erro quadrático ou soma das diferenças quadradas entre contornos de estruturas utilizando algoritmos de medida de similaridade entre intensidades de duas imagens(35,36). Nesse contexto, este trabalho apresenta uma abordagem singular em sistemas de recuperação de imagens médicas baseada em conteúdo, utilizando atributos de textura e o poder computacional da recente tecnologia de GC aplicada ao algoritmo de medida de similaridade baseado na soma das diferenças quadradas.
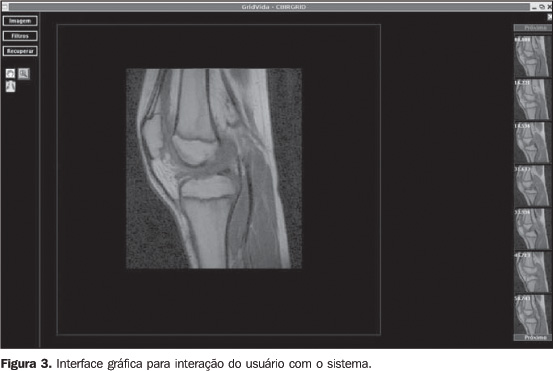
MATERIAIS E MÉTODOS O sistema desenvolvido no presente trabalho utilizou o sistema operacional GNU/Linux Debian e linguagem de programação Java 1.5, sendo o algoritmo de medida de similaridade (AMS) uma implementação do software Insight Toolkit(37). Para sua avaliação foi utilizada uma base heterogênea de imagens composta de 2.400 imagens de ressonância magnética de diversas regiões anatômicas, seqüências e planos de aquisição, e níveis de cinza variando entre 4.096 e 65.536. O sistema apresenta dois módulos de RIBC. O primeiro módulo utiliza análise de textura de segunda ordem (matriz de co-ocorrência) para realizar a classificação das imagens mais semelhantes de acordo com esta técnica. No segundo módulo é aplicado o AMS sobre as imagens selecionadas no primeiro módulo. Devido ao alto custo computacional do AMS, o segundo módulo é processado na GC do OurGrid. Este é uma GC cooperativa, aberta e de livre acesso, em que centros compartilham seus recursos computacionais ociosos e em reciprocidade, quando necessário, recebem recursos ociosos de outros centros. O OurGrid, atualmente, é composto por aproximadamente 500 máquinas. A interface do usuário com a GC é realizada pelo MyGrid 3.2 (OurGrid; Campina Grande, PB), que é o broker do OurGrid. Este é capaz de realizar a escolha dos recursos computacionais que vão ser utilizados na execução da aplicação, além de abstrair toda a complexidade da GC ao usuário; assim, o usuário utiliza a GC como se fosse um único computador(13). Todas as imagens da base de dados possuem um vetor de característica associado, que foi obtido pela matriz de co-ocorrência de níveis de cinza e seus atributos. A matriz de co-ocorrência seguiu as orientações em 0°, 45°, 90° e 135° e distância entre os elementos de imagens (pixels) igual a 1. Os atributos de textura utilizados na aplicação foram energia, entropia, momento da diferença inversa, penumbra, inércia, promenance, correlação e variância. A utilização de oito atributos de textura e quatro orientações angulares propiciou a criação de um vetor de característica de 32 dimensões. O sistema desenvolvido oferece uma interface gráfica (Figura 3) que permite ao especialista selecionar uma imagem de referência no padrão DICOM (digital imaging and communication in medicine), iniciando o primeiro módulo. Ao término do módulo as imagens são classificadas segundo o menor valor da distância euclidiana entre os vetores de características da imagem de referência e das imagens da base de dados.
O segundo módulo utiliza as 1.000 imagens mais similares segundo o primeiro módulo. Este módulo também necessita que o especialista defina em quantas tarefas o processamento do AMS será decomposto e distribuído na GC. Ou seja, qual a "granularidade" da aplicação. A granularidade está relacionada ao volume de imagens que será processado pelo AMS em cada computador da GC. O AMS utiliza transformações afins e interpolação linear buscando o mapeamento dos pontos homólogos entre duas imagens.
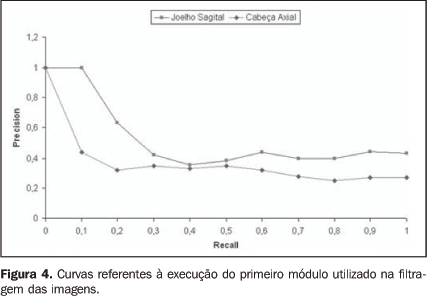
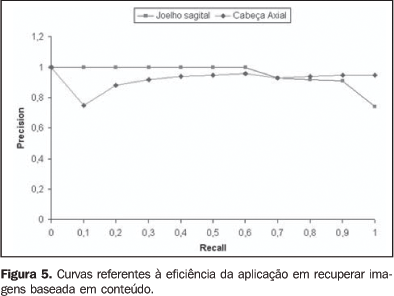
RESULTADOS Os resultados deste trabalho foram obtidos pela seleção de duas regiões anatômicas — joelho e cabeça — pertencentes à base de imagens. As imagens de joelho são ponderadas em T1 com plano de corte sagital e as imagens de cabeça são ponderadas em T2 e plano de corte axial. A base de dados possui um exame com 20 imagens de joelho no plano sagital e um exame com 40 imagens de cabeça no plano axial. Os experimentos foram repetidos três vezes escolhendo cortes diferentes dos exames descritos. As imagens foram classificadas como corretas quando a aplicação retornava imagens do mesmo plano e seqüência de aquisição das imagens de referência. O primeiro módulo classificou as imagens mais semelhantes segundo os atributos de textura. A média do tempo de processamento do primeiro módulo foi de 2,3 minutos, obtida pelo cálculo da distância euclidiana entre o vetor de característica de cada uma das 2.400 imagens da base de dados em relação ao vetor de característica da imagem de referência. Os algoritmos processados no computador local utilizaram processador Pentium 4 de 2.8 GHz com 1 Gbyte de memória. Para avaliarmos os resultados obtidos utilizamos precision (precisão) e recall (revocação), que são parâmetros normalmente utilizados para avaliar sistemas de recuperação de imagens baseada em conteúdo e recuperação de informação. Revocação indica a proporção de imagens relevantes na base de dados que foram recuperadas ao responder uma consulta. Precisão é a proporção das imagens recuperadas que são relevantes para a consulta(38). A Figura 4 ilustra as médias obtidas pela execução do primeiro módulo das curvas de revocação × precisão da distância euclidiana entre os vetores de características das imagens de referência em relação às imagens da base de dados. Este resultado permitiu avaliar a eficácia da RIBC utilizando textura na classificação das imagens mais semelhantes para o segundo módulo. Embora a média da precisão obtida nos experimentos seja de 0,54 (joelho sagital) e 0,40 (cabeça axial), ela é suficiente para filtrar as imagens que serão submetidas ao segundo módulo. No segundo módulo as imagens são processadas pelo AMS utilizando o processamento da GC. A RIBC utilizando o AMS produziu satisfatória precisão em ambos os casos estudados — 0,95 (joelho sagital) e 0,92 (cabeça axial) —, segundo as médias das curvas de revocação × precisão entre as imagens de referência e as classificadas pelo primeiro módulo (Figura 5).
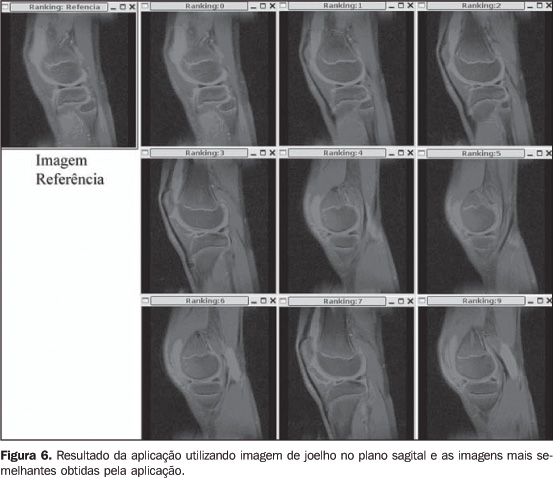
A Figura 6 ilustra a classificação das imagens mais semelhantes após a execução da aplicação. Devido a questões de espaço, somente as nove imagens mais semelhantes são ilustradas.
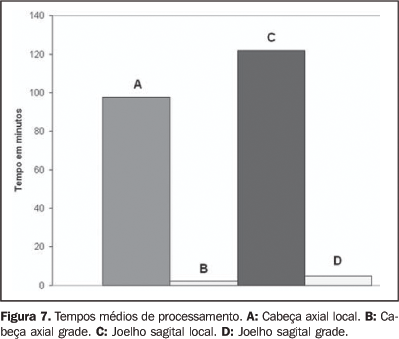
O alto custo computacional produzido pelo AMS foi amortizado pelo sistema de GC do OurGrid. Na média, o tempo de processamento do AMS aplicado aos experimentos utilizando 50 processadores da GC foi reduzido em 116,97 minutos para imagens de joelho e em 95,15 minutos para as imagens de cabeça, em relação aos tempos de processamento obtidos na máquina local (Figura 7).
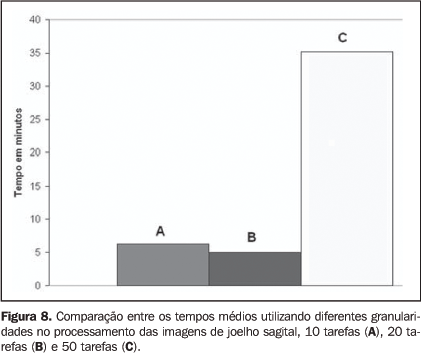
Neste experimento dividimos a aplicação em 20 tarefas composta por 50 imagens cada. As imagens foram compactadas antes de serem enviadas à GC e o tamanho médio dos arquivos com 50 imagens foi de 4 Mbytes. As imagens, ao serem enviadas à GC, possuem um arquivo de identificação único especificando o número da imagem e a qual tarefa ela pertence. O tempo médio do envio das imagens compactadas para as máquinas da GC foi de 22,2 segundos e o tempo médio de processamento das 50 imagens em cada máquina da GC foi de 11,45 segundos. O baixo tempo do envio das imagens é devido à maioria das tarefas ter sido executada nos computadores contidos na rede local. O OurGrid permitiu também que as bibliotecas necessárias a execução da aplicação fossem armazenadas nos computadores remotos, evitando a necessidade de reenvio de dados. O tempo médio dos experimentos foi também analisado alterando a granularidade da aplicação entre 10, 20 e 50 imagens por tarefa (Figura 8). A utilização do menor grão, ou seja, 50 imagens por tarefa, implicou uma maior divisão do volume total de imagens por tarefa. Logo, maior número de máquinas da GC foi requisitado, pois houve um aumento da quantidade de tarefas a serem processadas. A necessidade de alocar 50 máquinas da GC para executar a aplicação implicou a distribuição de tarefas para serem processadas fora da rede local. Assim, o tempo total da aplicação foi comprometido pelo tempo de transmissão de dados a computadores remotos.
Contudo, decompor a aplicação em tarefas maiores (dez tarefas no total), ou seja, maior grão, implicou a requisição de menos computadores e na transmissão de grandes arquivos, produzindo maior tempo de processamento executado por máquina. Logo, adotamos neste trabalho um número fixo e intermediário de 20 tarefas.
DISCUSSÃO A tecnologia de GC tem-se mostrado ferramenta promissora no processamento e armazenamento de grande volume de dados. Contudo, mais benefícios são esperados desta tecnologia, como o apresentado por Liu et al.(7), que utilizaram a arquitetura de GC para realizar cópias de segurança de imagens médicas em diversos sistemas PACS (picture archiving and communication system) pertencentes à GC. Este trabalho apresentou uma abordagem mista de técnicas de RIBC para classificar imagens semelhantes de diferentes regiões anatômicas e orientações utilizando o alto poder de processamento de GC. O sistema utilizou técnicas de RIBC baseadas na classificação de textura e medida de similaridade. A análise de textura apresenta boa aproximação para a percepção visual humana e tem sido utilizada em muitos sistemas de auxílio ao diagnóstico clínico(39,40). O valor médio da precisão da AT foi eficiente atuando como filtro inicial para o segundo módulo, apesar de relativamente baixa — 0,54 para joelho e 0,40 para cabeça. Uma possível solução para aumentar a eficiência dessa filtragem seria o desenvolvimento de métodos para detectar artefatos de movimento, já que informações de textura podem ser perdidas quando rotação, translação e escala são incluídas no espaço de características(29). A utilização do algoritmo de medida de similaridade da soma das diferenças quadradas aplicado ao segundo módulo apresentou valor médio de precisão bastante satisfatório — 0,95 para joelho e 0,92 para cabeça. O algoritmo foi capaz de recuperar imagens semelhantes de diferentes regiões anatômicas e orientações. A maioria dos artigos da literatura é restrita a determinada região anatômica, modalidade ou procedimento diagnóstico utilizando-se apenas vetores de características(41). No entanto, o custo computacional dos algoritmos de medida de similaridade, quando executados num único computador, é inaplicável ao diagnóstico auxiliado por computador. A GC viabiliza a utilização da técnica de medida por similaridade devido à capacidade de processar tarefas em paralelo nos diversos computadores que compõem a GC. Apesar de a GC utilizada neste trabalho possuir aproximadamente 500 computadores espalhados em mais de 20 locais, os experimentos com 10 e 20 tarefas foram processados nas máquinas da rede local, não comprometendo o tempo de execução da aplicação. Contudo, os experimentos com 50 tarefas necessitaram ser processados fora da rede local, logo, foram prejudicados pelo custo de transmissão dos dados. Nesses casos devemos avaliar o custo-benefício entre tempo de processamento e envio de dados. A tecnologia de GC ainda está nos primórdios em aplicações médicas, porém é bastante promissora e espera-se que, por meio de seus recursos, possibilite importantes avanços na área de informática aplicada à saúde. Para aprimorar os resultados obtidos, dois novos componentes estão em desenvolvimento: a medida de similaridade baseada na correlação cruzada e a segmentação automática de estruturas cerebrais. O algoritmo da correlação cruzada permitirá a busca em modalidades diferentes, o que é uma limitação do algoritmo de soma das diferenças quadradas. Outra limitação da soma das diferenças quadradas é a alta sensibilidade a pequenas quantidades de pixels que possuem grande diferença de intensidade entre duas imagens, como nos casos de injeção de contraste(35). O algoritmo de segmentação automática restringirá a recuperação de imagens a determinadas estruturas, permitindo buscas mais específicas do que as realizadas ao compararmos a imagem completa. A utilização de métodos distintos de forma integrada deverá proporcionar uma melhor discriminação entre as imagens(42). Agradecimentos Ao Projeto GridVida, ao Laboratório de Sistemas Distribuídos da Universidade Federal de Campina Grande (UFCG) e ao Centro de Ciências das Imagens e Física Médica (CCIFM) da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP-USP).
REFERÊNCIAS 1. Montagnat J, Breton V, Magnin IE. Using technologies to face medical image analysis challenges. Proceedings of the IEEE CCGrid03 2003, Tokyo, Japan. [ ] 2. Montagnat J, Breton V, Magnin IE. Partitioning medical image databases for content-based queries on a Grid. Methods Inform Med 2005;44: 154–160. [ ] 3. Azevedo-Marques PM. Diagnóstico auxiliado por computador na radiologia. Radiol Bras 2001;34: 285–293. [ ] 4. HealthGrid, HealthGrid White Paper. [Acessado em: 10/10/2006]. Disponível em: http://www. heathgrid.org [ ] 5. Montagnat J, Bellet F, Benoit-Catin H, et al. Medical images simulation, storage, and processing on the European DataGrid testbed. J Grid Comput 2004;2:387–400. [ ] 6. Breton V, Blanchet C, Legré Y, Maigne L, Montagnat J. Grid technology for biomedical applications. Lecture Notes in Computed Science 2005; 204–218. [ ] 7. Liu BJ, Zhou MZ, Documet J. Utilizing data Grid architecture for the backup and recovery of clinical image data. Comput Med Imaging Graph 2005;29:95–102. [ ] 8. Foster I, Kesselman C, Tuecke S. The anatomy of the Grid: enabling scalable virtual organizations. International Journal of High Performance Computing Applications 2001;15:200–222. [ ] 9. Foster I, Kesselman C. The Grid 2: blueprint for a new computing infrastructure. San Francisco, CA: Morgan Kaufmann Publishers, 2004. [ ] 10. Foster I, Kesselman C. Globus: a metacomputing infrastructure toolkit. International Journal of Supercomputing Applications 1997;11:115–128. [ ] 11. Grimshaw AS, Wulf WA. The legion vision of a worldwide virtual computer. Communications of the ACM 1997;40:39–45. [ ] 12. Condor. [Acessado em: 13/9/2006]. Disponível em: http://www.cs.wisc.edu/condor [ ] 13. Cirne W, Brasileiro F, Andrade N, et al. Labs of the World, Unite!!! UFCG/DSC Technical Report 07/2005;1–12. [ ] 14. de Roure D, Baker M, Jennings NR, Shadbolt N. The evolution of the Grid. In: Berman F, Fox G, Hey AJG, editors. Grid computing – making the global infrastructure a reality. New York, NY: Wiley, 2003;65–100. [ ] 15. Foster I. The Grid: computing without bounds. Scientific American April 2003,228:80–85. [ ] 16. Grid Café. [Acessado em: 29/8/2006]. Disponível em: http://gridcafe.web.cern.ch/gridcafe [ ] 17. Montagnat J, Duque H, Pierson JM, Breton V, Brunie L, Magnin IE. Medical image content-based queries using the Grid. Proceedings of the First European HealthGrid Conference 2004, Lyon, France. [ ] 18. Rahman M, Wang T, Desai B. Medical image retrieval and registration: towards computer assisted diagnostic approach. In: IDEAS Workshop on Medical Information Systems: The Digital Hospital (IDEAS-DH'04), 2004. Washington, DC: IEEE Computer Society, 2004;78–89. [ ] 19. Güld MO, Kohnen M, Keysers D. Quality of DICOM header information for image categorization. Proceedings of the International Symposium on Medical Imaging 2002, San Diego, CA. [ ] 20. Tagare HD, Jaffe C, Duncan J. Medical image databases: a content-based retrieval approach. J Am Med Inform Assoc 1997;4:184–198. [ ] 21. Traina Júnior C, Traina AJM, Santos RR, Senzako EJ. A support system for content-based medical image retrieval in object oriented databases. J Med Syst 1997;21:339–352. [ ] 22. Rosset A, Ratib O, Valle J. Integration of a multimedia teaching and reference database in a PACS environment. RadioGraphics 2002;22:1567–1577. [ ] 23. Petrakis EGM. Content-based retrieval of medical images. Int J Comput Res 2002;11:171–182. [ ] 24. Müller H, Michoux N, Bandon D, Geissbuhler A. A review of content-based image retrieval systems in medical applications – clinical benefits and future directions. Int J Med Inform 2004;73: 1–23. [ ] 25. Azevedo-Marques P, Honda MH, Rodrigues JAH, et al. Recuperação de imagem baseada em conteúdo: uso de atributos de textura para caracterização de microcalcificações mamográficas. Radiol Bras 2002;35:93–98. [ ] 26. Haralick RM, Shanmuga K, Dinstein I. Textural features for image classification. IEEE Trans Syst Man Cybern 1973;SMC3:610–621. [ ] 27. Walker RF, Jackway P, Longstaff ID. Improving co-occurrence matrix feature discrimination. In: Proc DICTA '95, 3rd Conference on Digital Image Computing: Techniques and Application 1995;643–648. [ ] 28. McLean GF. Vector quantization for texture classification. IEEE Trans Syst Cybern 1993;23:637–644. [ ] 29. Freeborough PA, Fox NC. MR image texture analysis applied to the diagnosis and tracking of Alzheimer's disease. IEEE Trans Med Imaging 1998;17:475–479. [ ] 30. Mathias JM, Tofts PS, Losseff NA. Texture analysis of spinal cord pathology in multiple sclerosis. Magn Reson Med 1999;42:929–935. [ ] 31. Materka A, Strzelecki M. Texture analysis methods – a review. In: COST B11 Report. Lodz, Poland: Technical University of Lodz, Institute of Electronics, 1998. [ ] 32. Konak ES. A content-based image retrieval system for texture and color queries. (M.Sc. degree thesis). Ankara, Turkey: Department of Computer Engineering and Institute of Engineering and Science, Bilkent University, 2002. [ ] 33. Sharma M, Singh S. Evaluation of texture methods for image analysis. 7th Australian and New Zealand Intelligent Information System Conference. Perth, Australia, 2001;117–121. [ ] 34. Veltkamp RC, Hagedoorn M. State-of-the-art in shape matching. In: Lew M, editor. Principles of visual information retrieval. London: Springer-Verlag, 2000;87–119. [ ] 35. Hajnal JV, Hill DLG, Hawkes DJ. Medical image registration. In: Neuman MR, editor. Biomedical engineering. Boca Raton, FL: CRC Press, 2001. [ ] 36. Yoo TS. Insight into images: principles and practice for segmentation, registration, and image analysis. Wellesley, MA: AK Peters, 2004. [ ] 37. InsightToolkit. [Acessado em: 8/10/2006]. Disponível em: http://www.itk.org [ ] 38. Bueno JM. Suporte à recuperação de imagens médicas baseada em conteúdo através de histogramas métricos. São Carlos, SP: Instituto de Ciências Matemáticas e de Computação, Universidade de São Paulo, 2002. [ ] 39. Shyu CR, Bradley CE, Kak AC, Kosaka A, Aisen AM, Broderick LS. ASSERT: a physician-in-the-loop content-based retrieval system for HRCT image databases. Computer Vision and Image Understanding 1999;75:111–132. [ ] 40. Kuo WJ, Chang RF, Lee CC, Moon WK, Chen DR. Retrieval technique for the diagnosis of solid breast tumors on sonogram. Ultrasound Med Biol 2002;28:903–909. [ ] 41. Lehmann TM, Güld MO, Thies O, et al. Content-based image retrieval in medical applications. Methods Inform Med 2004;43:354–361. [ ] 42. Traina AJM, Traina C, Bueno JM, Chino FJT, Azevedo-Marques P. Efficient content-based image retrieval through metric histograms. World Wide Web J 2003;6:157–185. [ ]
Recebido para publicação em 21/11/2005. Aceito, após revisão, em 23/10/2006.
* Trabalho realizado no Laboratório de Sistemas Distribuídos do Departamento de Sistemas e Computação da Universidade Federal de Campina Grande (UFCG), Campina Grande, PB, Brasil. |
|
Av. Paulista, 37 - 7° andar - Conj. 71 - CEP 01311-902 - São Paulo - SP - Brazil - Phone: (11) 3372-4544 - Fax: (11) 3372-4554